Une ontologie pour considérer les multiples dimensions des systèmes agroalimentaires dans une approche circulaire et plus durable
Résumé
Face aux défis de la transition agroalimentaire et aux exigences de durabilité, il est essentiel de combiner des données portant sur de multiples dimensions. Aujourd’hui, ces données sont souvent dispersées et difficiles à interconnecter, ce qui limite leur exploitation et leur réutilisation. La gestion et l’ouverture des données scientifiques deviennent essentielles afin de structurer et partager efficacement les connaissances. La construction de l’ontologie PO²/TransformON répond à ce besoin en proposant une approche fondée sur les principes FAIR (Facile à trouver, Accessible, Interopérable et Réutilisable) et le Web sémantique. L’ontologie vise à organiser et modéliser les données liées aux procédés de transformation et à la caractérisation des produits alimentaires et non alimentaires. L’objectif est de faciliter leur partage et leur exploitation par la communauté scientifique. Dans cette optique, le processus de FAIRification s’appuie sur le développement des solutions logicielles PO² Manager et SPO²Q. Ces solutions permettent de structurer les informations, dès leur acquisition, pour les représenter sous forme de graphes et de les requêter à l’aide des standards du Web sémantique RDF, OWL et SPARQL. L’intégration de ces données qui répondent aux principes FAIR dans des entrepôts ouverts, comme Recherche Data Gouv, favorise leur réutilisation et leur valorisation. À terme, cette démarche ouvre de nouvelles perspectives, notamment en intelligence artificielle et en modélisation prédictive pour l’aide à la décision. L’enrichissement des ontologies et l’interopérabilité accrue des systèmes d’information faciliteront la gestion durable des ressources et l’innovation dans les domaines agroalimentaire et environnemental.
Glossaire1
FAIRification (ou FAIRisation) : Ensemble des processus qui amènent à produire des données FAIR.
Format JSON (« Javascript Object Notation ») : Format texte de données, dérivé de la notation des objets du langage JavaScript, permettant la représentation et la transmission d’informations structurées.
Format TSV (« Tab-Separated Values ») : Format texte ouvert représentant des données tabulaires sous forme de valeurs séparées par des tabulations.
Intelligence artificielle (IA) : Processus qui repose sur la création et l’exécution d’algorithmes dans un environnement informatique dynamique. Il vise à concevoir des systèmes capables de raisonner, d’apprendre et d’agir de manière autonome. Ces systèmes réalisent des tâches qui nécessitent normalement l’intelligence humaine ou traitent des volumes de données trop importants pour être analysés par un humain.
Interopérabilité sémantique : Capacité des systèmes informatiques à échanger des données dont la signification n’est pas ambiguë. Cela est possible grâce à l’utilisation de vocabulaires contrôlés qui sont partagés au sein d’une communauté de pratique.
Principes FAIR : Ensemble de principes directeurs pour gérer les données de la recherche visant à les rendre faciles à trouver, accessibles, interopérables et réutilisables par l’Homme et la machine.
Resource Description Framework (RDF) : Modèle de description de ressources Web et de leurs métadonnées, afin de permettre le traitement automatique de ces descriptions.
Science ouverte : Mouvement qui vise à partager les connaissances et productions scientifiques, non seulement avec l’ensemble de la communauté scientifique, mais aussi avec tous les acteurs de la société.
Simple Knowledge Organization System (SKOS) : Modèle de données utilisé pour représenter de manière standardisée des systèmes d’organisation des connaissances simples, comme des vocabulaires contrôlés, thésaurus ou dictionnaires.
SPARQL Query Language : Langage de requête et protocole qui permettent de rechercher, d’ajouter, de modifier ou de supprimer des données RDF (Resource Description Framework) disponibles à travers Internet.
Uniform Resource Identifier (URI, identifiant uniforme de ressource) : Chaîne de caractères identifiant, de manière unique et non équivoque, une ressource sur un réseau, notamment une ressource Web.
One Health (« Une seule santé ») : Concept qui vise à prendre en compte les interdépendances entre l’état de santé des différents domaines du vivant (des écosystèmes à l’Homme). Considérer ces interdépendances permet de renouveler l’analyse de la durabilité des systèmes agroalimentaires.
Web (« World Wide Web ») : Système d’information reposant sur des hyperliens, également appelés liens hypertextes, permettant d’accéder à des contenus et des services via le réseau internet.
Web Ontology Language (OWL) : Langage de représentation de connaissances du Web sémantique permettant de définir des ontologies en mettant en œuvre une sémantique formelle basée sur certaines logiques de description.
Web sémantique : Extension du Web standardisée par le World Wide Web Consortium (W3C) s’appuyant sur le modèle Resource Description Framework (RDF) et permettant aux données d’être partagées sur le Web.
Introduction
Contexte général
Pour répondre aux enjeux de santé globale sous le concept « One Health » (World Health Organization : WHO, 2023
Le « FAIR-by-design » est une approche qui consiste à appliquer les principes FAIR dès la phase d’acquisition de la donnée et tout au long de son cycle de vie (Produire des données FAIR, INRAE, DipSO
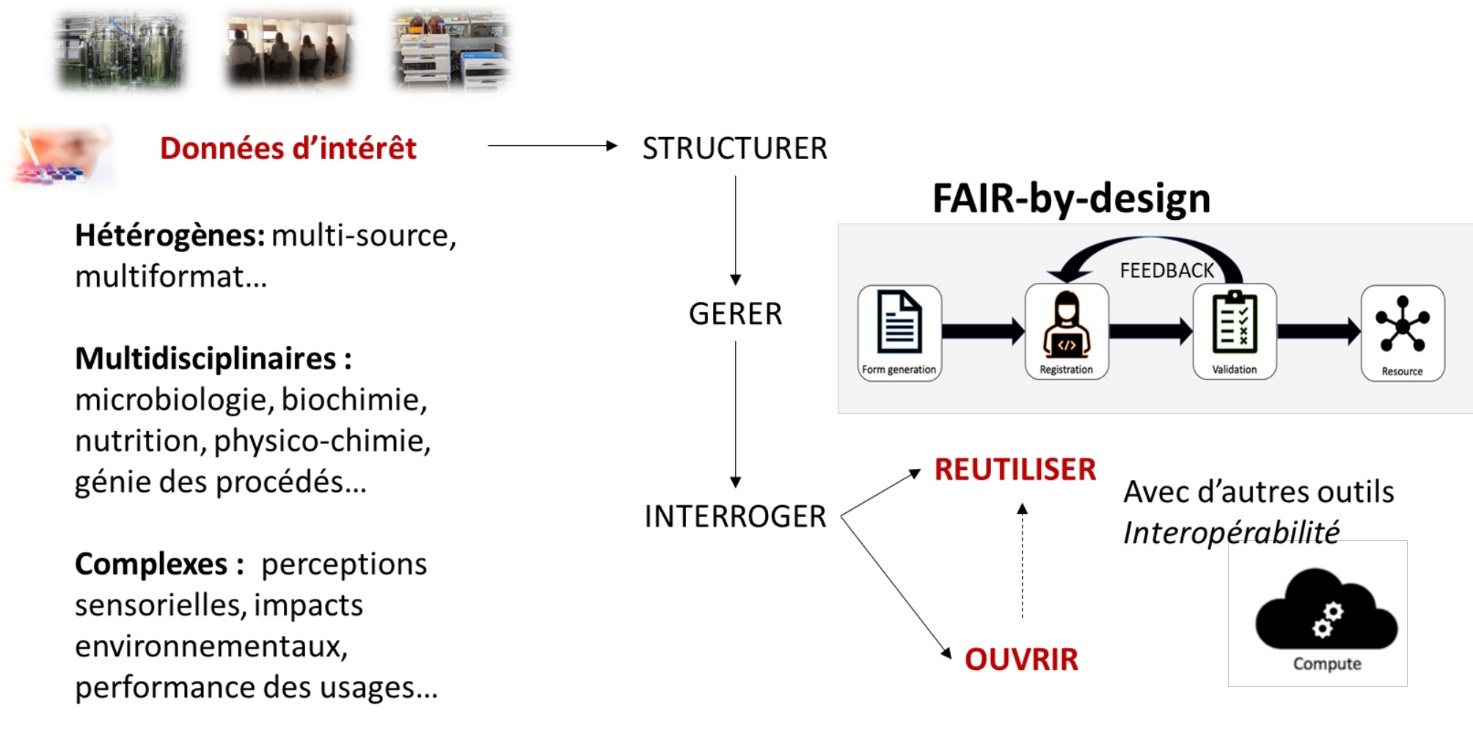
Figure 1. Approche « FAIR-by-design » de la structuration des données
Qu’est-ce qu’une ontologie et à quoi sert-elle ?
Les ontologies font partie des techniques de l’intelligence artificielle et rendent possible la formalisation des connaissances à partir des données. Grâce à l’explicitation du contexte d’obtention et du type des données, et en s’appuyant sur un vocabulaire consensuel et partagé par les experts pour décrire les différents concepts, elles facilitent le dialogue entre communautés scientifiques et permettent l’interopérabilité sémantique entre des systèmes d’information. Plus précisément, une ontologie est un modèle de connaissances qui permet de représenter un ensemble de concepts ou types d’entités représentés par des classes, avec des relations et des propriétés. Chaque concept permet de décrire une entité matérielle, une notion abstraite ou un type d’évènement. Les relations entre concepts permettent d’exprimer des connaissances du domaine, de faire des catégorisations (appelées classes de concepts) et du raisonnement. Cette représentation symbolique rend possible la création de graphes de connaissances exploitables par des ordinateurs grâce aux technologies du Web sémantique : RDF, OWL, SPARQL. Le graphe de connaissances est représenté à l’aide du format RDF (Resource Description Framework) permettant de relier les concepts avec des identifiants uniques appelés URI (Uniform Resource Identifier). La sémantique formelle du graphe est représentée avec le langage OWL (Web Ontology Language). Il existe différents types d’ontologies selon leur niveau de formalisme et leur généricité ; ces ontologies peuvent être combinées et spécialisées pour les besoins des applications ou des domaines de connaissances particuliers (voir la page https://vocabulaires-ouverts.inrae.fr/types-de-vocabulaires-et-usages pour plus d’information).
Problématique
Pour nos cas d’usage, l’ontologie a pour objectif de décrire et de structurer des données potentiellement hétérogènes et issues de multiples sources, en lien avec les enjeux de recherche suivants :
- réduction des pertes alimentaires et valorisation des déchets ou effluents agroalimentaires ;
- production d’énergie, de matériaux biosourcés, de bioproduits ou de molécules d’intérêt produites à partir de la biomasse ;
- évaluation et amélioration de la qualité fonctionnelle, nutritionnelle, sanitaire, environnementale et sensorielle des aliments ;
- écoconception de procédés et évaluation des impacts environnementaux des systèmes agroalimentaires et de bioraffinerie.
Solution apportée
La solution qui a été développée se base sur le modèle générique appelé « Process and Observation Ontology » (PO²) permettant la description d’un processus générique de transformation et de caractérisation (Ibanescu et al., 2016). Ce modèle a été étendu, en réutilisant des concepts d’autres ontologies standard, et spécialisé, avec la création d’un vocabulaire spécifique au domaine de l’ingénierie des aliments et des bioproduits, pour construire l’ontologie de domaine TransformON (Weber et al., 2023).
L’ontologie PO²/TransformON couvre le domaine de l’ingénierie des aliments et des bioproduits depuis les matières premières jusqu’aux produits finis, incluant les déchets d’origine végétale, animale ou humaine. Ce domaine englobe la transformation et la caractérisation de tous les produits issus de la biomasse, obtenus par des procédés alimentaires ou non alimentaires (génie chimique, génie biologique ou biotechnologies). Les champs d’application portent sur la bioéconomie (l’économie de la photosynthèse et plus largement du vivant) incluant l’alimentation, comme illustré sur la Figure 2. Ainsi, le périmètre couvre les systèmes alimentaires et inclut les aspects socio-économiques, environnementaux, nutritionnels en lien avec la santé et le bien-être des consommateurs, dans une conception systémique incluant la valorisation des co-produits et résidus.
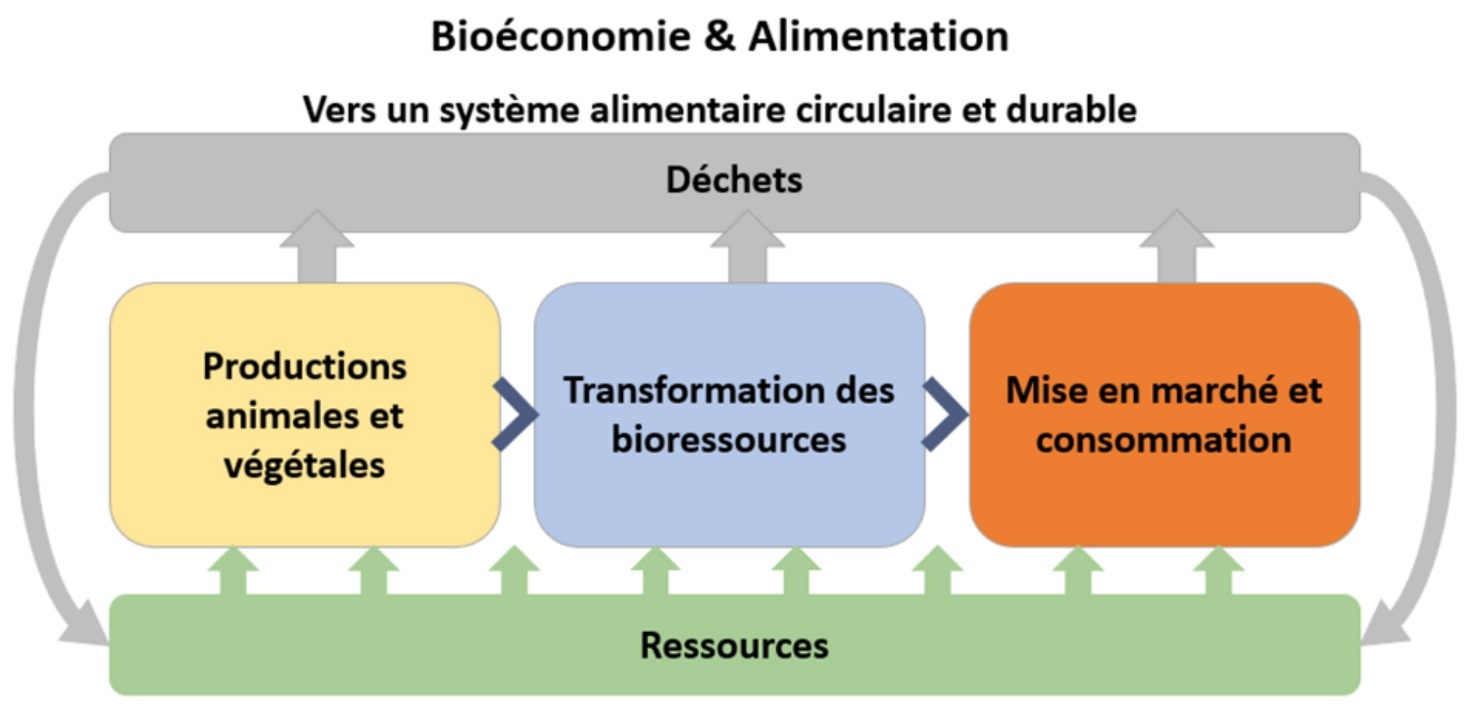
Figure 2. Domaine couvert par l’ontologie PO²/TransformON : bioéconomie et alimentation
Dans les sections suivantes, nous présentons la démarche de construction de l’ontologie, le modèle utilisé et les hiérarchies contenant le vocabulaire spécialisé sur le domaine. Nous montrons ensuite comment utiliser cette ontologie pour structurer et exploiter les données à l’aide des outils développés, avant de déployer deux exemples d’application sur les domaines alimentaire et non alimentaire.
Démarche de construction de l’ontologie PO²/TransformON
La construction de l’ontologie PO²/TransformON utilise un des scénarios de construction d’ontologie où l’on réutilise des ressources existantes, et s’appuie sur la méthodologie Linked Open Terms (Poveda-Villalón et al., 2022).
Pour développer l’ontologie de domaine et collecter les concepts et termes nécessaires à la représentation des connaissances, nous avons combiné deux approches complémentaires ; premièrement, nous avons suivi une démarche ascendante (« bottom-up ») avec des cas d’utilisation et des ensembles de données existants (approche guidée par les données) ; et deuxièmement, nous avons suivi une démarche descendante pour sélectionner les ressources appropriées pour construire la hiérarchie sur le domaine (« top-down »).
Lors de la phase « bottom-up », la constitution du vocabulaire s’est appuyée sur la sélection de termes préférentiels destinés à décrire les concepts, et des synonymes collectés à partir des cas d’utilisation, avec l’aide d’experts du domaine et de recherches bibliographiques. Pour la recherche d’ontologies existantes, lors de la phase « top-down », nous avons utilisé des services spécifiques tels que Ontology Lookup Service (OLS, https://www.ebi.ac.uk/ols/index), un référentiel d’ontologies biomédicales, et AgroPortal (https://agroportal.lirmm.fr), le portail de ressources sémantiques en sciences agronomiques et alimentaires. Par rapport au champ d’application, nous avons sélectionné FoodOn (https://foodon.org) et l’ontologie Compositional Dietary Nutrition Ontology (CDNO, https://www.ebi.ac.uk/ols4/ontologies/cdno) comme ressources principales en ce qui concerne l’alimentation (Dooley et al., 2018 ; Andrés-Hernandez et al., 2022). Les ontologies Ontology for Biomedical Investigations (OBI, https://obi-ontology.org), Chemical Methods Ontology (CHMO, https://www.ebi.ac.uk/ols/ontologies/chmo) et Chemical Entities of Biological Interest (ChEBI, https://www.ebi.ac.uk/chebi) ont été sélectionnées pour la description des méthodes de caractérisation et de certains composants chimiques.
Afin de couvrir l’ensemble du domaine visé, nous avons également pris en considération d’autres ressources telles que FoodEx2 (https://www.efsa.europa.eu/fr/data/data-standardisation), le référentiel européen établi par l’EFSA pour les aliments destinés à l’alimentation humaine et animale, ainsi que le Catalogue européen des déchets, une liste hiérarchique de catégories de déchets établie par la Commission européenne (décision 2000/532/CE
Des alignements ont ensuite été réalisés entre PO²/TransformON et une version sémantique de FoodEx2 mise à disposition par l’agence britannique Food Standards Agency (https://data.food.gov.uk/codes/ui/about-foodtypes), c’est-à-dire une version en RDF hébergée sur un serveur qui permet d’afficher des pages web à partir des URI grâce au mécanisme de déréférencement. Ces alignements se matérialisent par des relations de correspondance qui fonctionnent comme des liens hypertextes et qui permettent de naviguer entre les ressources ainsi reliées entre elles. Un second travail d’alignement est en cours avec le Thésaurus INRAE (https://vocabulaires-ouverts.inrae.fr/a-propos-du-thesaurus-inrae) pour permettre l’interopérabilité entre les domaines thématiques ou les communautés disciplinaires au sein de l’institut. Le Thésaurus est lui-même en partie aligné avec d’autres vocabulaires et constitue une ressource centrale pour les systèmes d’information à INRAE (Aubin et al., 2025). Enfin, des alignements sont prévus avec l’ontologie FoodOn qui contient des concepts relatifs aux aliments ou plus largement à l’alimentation, issus de différentes ressources internationales, comme illustré par la Figure 3.
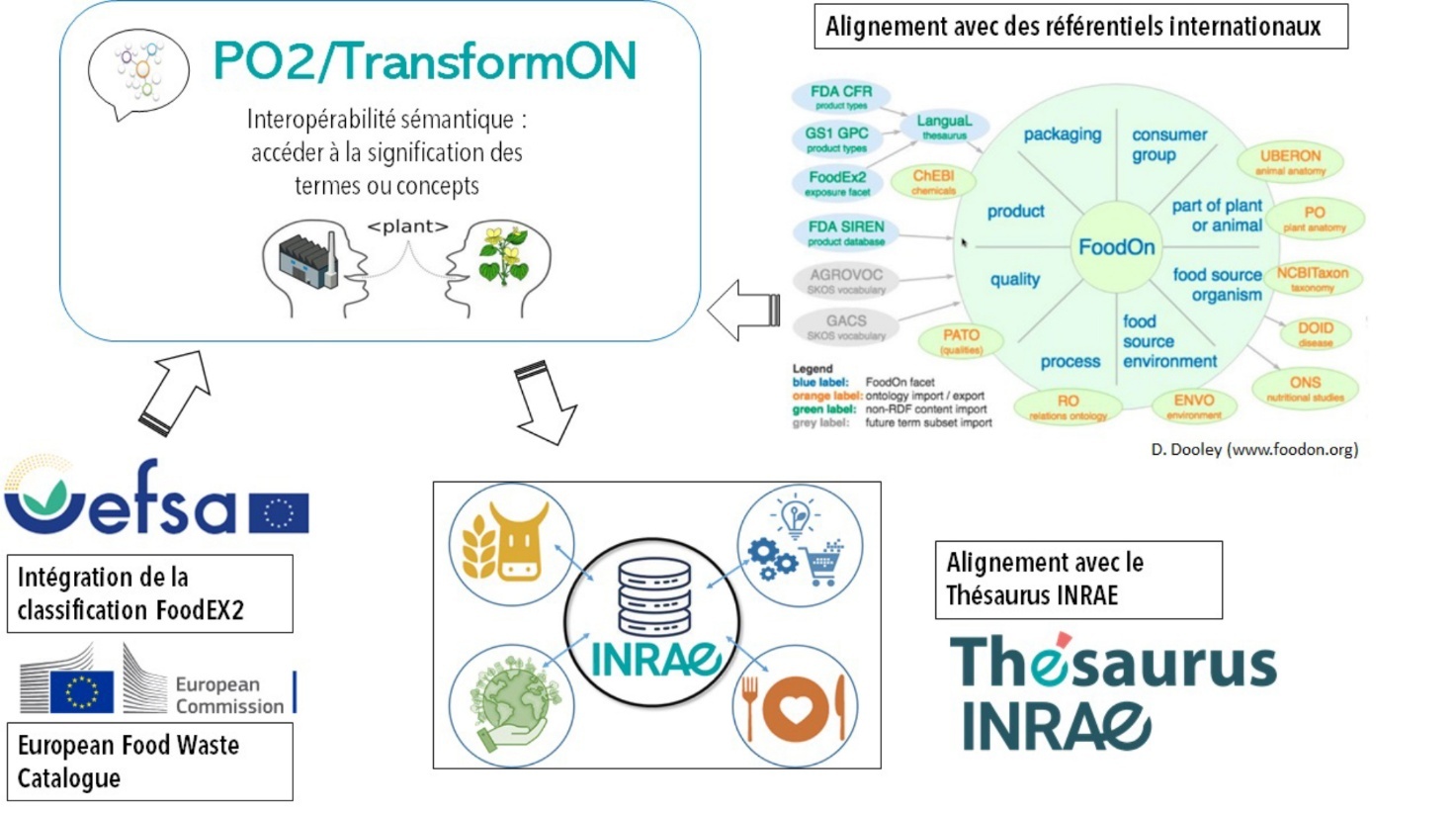
Figure 3. Stratégie d’intégration et d’alignement de PO²/TransformON
Le modèle Process and Observation Ontology (PO²)
Le modèle générique PO² permet la description des procédés et les observations associées. Il réutilise diverses ontologies ou schémas standard existants comme indiqué dans le Tableau 1 : SOSA/SSN pour représenter les observations, Time Ontology pour représenter les relations temporelles, QUDT pour les unités, DCAT et PROV-O pour les informations relatives à la provenance des données, et des balises de données structurées de Schema.org. Par ailleurs, le modèle PO² est cohérent avec celui défini par le consortium OBO Foundry sur lequel se basent de nombreuses ontologies dans le domaine des sciences du vivant (Dooley et al., 2024) et reprend des concepts des ontologies BFO et IAO (voir Tableau 1).
Nom de la ressource | Objet/domaine | URL de la ressource |
SOSA/SSN : Semantic Sensor Network Ontology | Observations, capteurs, actionneurs, échantillons | |
Time Ontology in OWL | Temporalité | |
BFO : Basic Formal Ontology | Ontologie fondationnelle des ontologies OBO | |
IAO : Information Artifact Ontology | Représentation de l’information | |
QUDT : Quantities, Units, Dimensions and Types | Unités de mesure, quantités, dimensions, types de données | |
DCAT : Data Catalog Vocabulary | Description de jeux de données | |
PROV-O : The PROV Ontology | Information de provenance | |
Schema.org | Balises de données structurées |
Vue simplifiée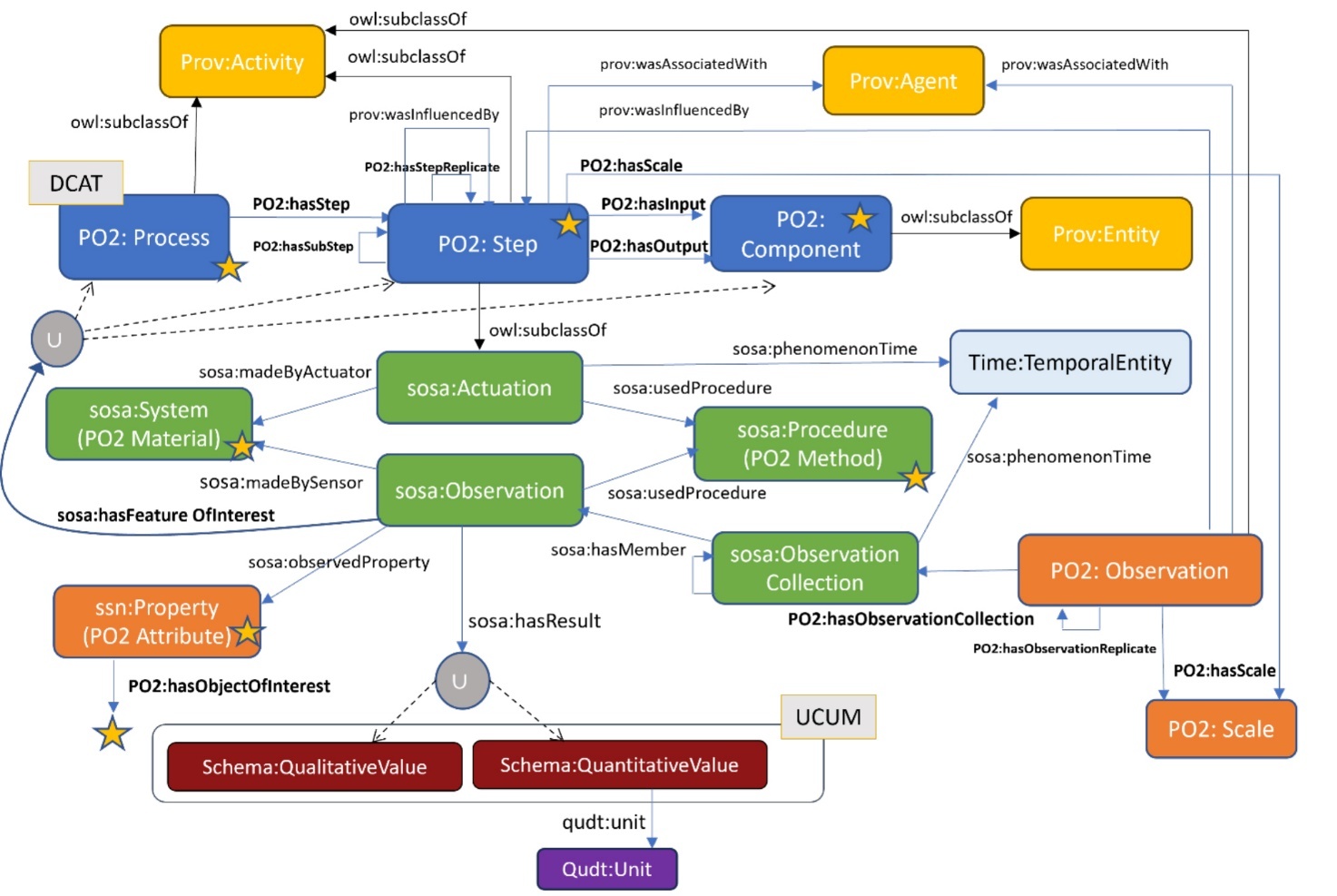
Figure 4. Représentation synthétique du modèle PO² V2.4
La Figure 4 montre le modèle PO² de façon synthétique. Les éléments en bleu foncé constituent la partie « Processus ». La partie « Observation » provient principalement de SOSA (éléments en vert) et est complétée par des concepts PO² ou SSN (éléments en orange). Un agent PROV-O peut être associé aux actuations et aux observations définies comme classes de prov:Activity (éléments en jaune). Les résultats des observations sont des valeurs quantitatives ou qualitatives (éléments en gris), associés à des unités (l’élément en violet) avec des codes UCUM dans le cas des valeurs quantitatives. Le symbole U indique une union de classes. Les étoiles indiquent les objets d’intérêt qui peuvent être associés à une caractéristique observée. Des métadonnées issues de DCAT donnent des informations sur la provenance des données. Les flèches en pointillés concernent des instances.
- La partie « Processus » décrit une succession d’étapes (PO²/Step) et les composants ou compositions d’entrée et de sortie de chacune d’elles (PO²/Component). Une étape représente une action ou opération réalisée au moyen d’un concept sosa:Actuation auquel est associé un intervalle de temps défini par Time:TemporalEntity .
- La partie « Observation » réutilise les concepts de l’ontologie SOSA. Chaque sosa:Observation est réalisée à l’aide d’un matériel (PO²:Material qui est le concept sosa:System) en suivant certaines méthodes (PO²/Method qui est le concept sosa:Procedure). Une PO²/Observation (concept en orange) est une collection de sosa:Observations qui peuvent porter sur un procédé complet (PO²/Process), une étape (PO²/Step), ou une composition (PO²/Component). Une observation contient également les paramètres de contrôle associés aux équipements ou aux méthodes. Le concept PO²/Scale permet de décrire les échelles de mesure associées aux observations et les échelles liées aux procédés de transformation.
- La partie « Résultat » traite des caractéristiques observées, mesurées ou calculées (PO²/Attribute) qui sont des SSN:Property. Ces caractéristiques sont associées à d’autres concepts à l’aide de la relation PO²/hasObjectOfInterest qui précise quel est l’objet d’intérêt. Les valeurs qualitatives ou quantitatives associées aux résultats sont décrites à l’aide du standard Schema.org. Les unités de mesure associées aux valeurs quantitatives sont liées à l’ontologie QUDT grâce au standard Unified Code for Units of Measure (UCUM, https://ucum.org), qui permet la normalisation des unités de mesure et des conversions de valeur selon le système international.
Focus sur le contenu de PO²/TransformON
Lors de la construction d’une ontologie de domaine basée sur le modèle générique PO², sept concepts « cœurs » doivent être spécialisés pour construire des hiérarchies de classes de concepts décrivant le domaine : PO²/Process, PO²/Step, PO²/Component, PO²/Material, PO²/Method, PO²/Attribute et PO²/Scale.
La Figure 5 montre les niveaux supérieurs de l’ontologie du domaine PO²/TransformON dans sa version 17.0 ainsi que les principales métriques associées.
Seuls les deux premiers niveaux sont indiqués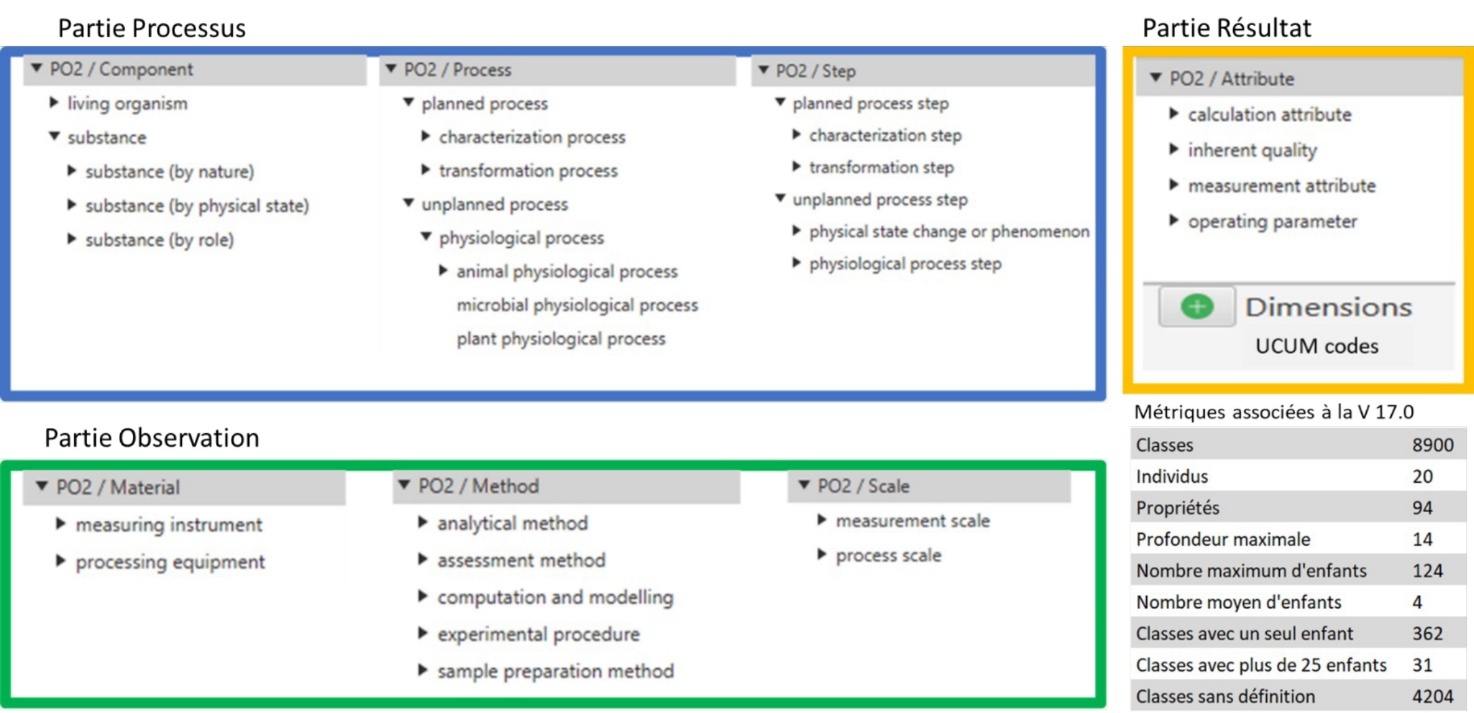
Figure 5. Représentation des hiérarchies de PO²/TransformON
L’ensemble des hiérarchies inclut 8 900 classes définies au format OWL2. Le Tableau 2 indique le nombre de classes pour chaque branche de l’ontologie PO²/TransformON et rappelle les ressources utilisées pour construire les hiérarchies.
Hiérarchie de concepts | Nombre de classes | Ressources utilisées |
PO² Attribute | 1 533 classes | FoodEx2 |
PO² Component à Living organisms à Substance à Substance by nature à Substance by physical state à Substance by role à Dietary substance à Food à Feed à Dietary constituents à Non-food substance | 5 820 classes 65 classes 376 classes 69 classes 5 305 classes 4 242 classes 759 classes 49 classes 252 classes |
NCBI FoodEx2, CheBI FoodEx2 FoodEx2, FoodOn FoodEx2 CNDO CheBI European Waste Catalogue Référentiel SINOE |
PO² Material | 455 classes | OBI, CHMO |
PO² Method | 491 classes | OBI, CHMO, IUPAC |
PO² Process | 36 classes | FoodEx2, FoodOn |
PO² Scale | 24 classes | |
PO² Step | 545 classes | OBI, FoodEx2 |
La hiérarchie de concepts peut être représentée séparément à l’aide du format SKOS (Simple Knowledge Organization System). Le vocabulaire défini au format SKOS constitue la partie terminologique de l’ontologie, et on parle alors de termino-ontologie. SKOS permet de gérer les traductions (termes en anglais et en français), la synonymie et les relations de correspondance entre concepts (voir https://vocabulaires-ouverts.inrae.fr/skos-un-standard-pour-une-ressource-semantique-simple-et-fair).
Partie « Processus » (Component, Process, Step)
La partie « Processus » contient trois hiérarchies : PO²/Component, PO²/Process, PO²/Step (Figure 6).
PO²/Component
Lors de la construction de la branche PO²/Component, nous avons distingué les organismes vivants des substances telles que les différents types de biomasse, l’énergie, les composés chimiques, etc. Les organismes vivants sont les sources biologiques des matières premières entrant dans les processus de transformation après récolte ou abattage. La classe « Living organism » regroupe les catégories d’organismes vivants selon les trois grands niveaux de la taxonomie reconnus actuellement (Archaea, Bacteria, Eukarya). Seules les classes de haut rang taxonomique ont été créées (soit 65 classes) et nous ajouterons les organismes au fur et à mesure des besoins d’annotation. Hormis pour les microorganismes listés par taxons (genre, espèce, voire souche), les organismes sont désignés par leurs noms communs (noms vernaculaires). Des alignements avec des ressources taxonomiques seront effectués pour relier ces noms communs à des taxons scientifiques, à l’aide de la taxonomie NCBI (https://www.ncbi.nlm.nih.gov/taxonomy).
Trois points de vue permettent de décrire plus précisément les substances : leur nature (biologique ou physico-chimique), leur état physique (solide, liquide, etc.) ou encore leur usage (alimentaire ou non alimentaire).
La hiérarchie « Substances alimentaires » comporte les sous-classes « Food », « Feed » et « Dietary constituents ». La branche « Food » est basée sur la hiérarchie principale proposée dans FoodEx2. Elle comprend 4 242 classes directement importées de FoodEx2. Certaines classes intermédiaires ont été ajoutées pour faciliter les futurs alignements avec FoodOn, par exemple pour distinguer les animaux vertébrés des invertébrés, ou les substances animales comme le lait cru ou les œufs. La branche « Feed » comprend 759 classes importées de FoodEx2 mais deux sous-classes parent ont été créées dans la hiérarchie : l’une pour regrouper toutes les sources primaires d’aliments pour animaux et l’autre pour regrouper les aliments composés pour animaux pour reprendre la même logique que celle de la branche « Food ». Les constituants alimentaires correspondants aux éléments nutritionnels sont listés dans la branche « Dietary constituents » qui comprend 49 classes. Les constituants alimentaires seront alignés avec l’ontologie CDNO et sur CheBi (Andrés-Hernández et al., 2022).
Seuls les trois premiers niveaux de la hiérarchie sont montrés
Figure 6. Détail des hiérarchies PO²/Component, PO²/Process et PO²/Step
Nous avons basé la construction de PO²/TransformON sur FoodEx2 en reprenant toutes les catégories des denrées alimentaires (« Food ») et des aliments pour animaux (« Feed ») présentes dans la hiérarchie principale de FoodEx2 (« Matrix hierarchy ») car FoodEx2 est un système de classification et de description des aliments qui fait référence au niveau européen pour l’annotation des bases de données de contamination et de consommation alimentaire et l’échange de ces données. Il est d’ailleurs utilisé par l’Anses pour décrire les aliments présents dans ses bases de données de consommation (études Inca, https://www.anses.fr/fr/content/les-etudes-inca) et de contamination (base Contamine). L’Anses utilisera également FoodEx2 pour l’annotation de la base de données de composition nutritionnelle CIQUAL (https://ciqual.anses.fr). La classification de FoodEx2 est bien adaptée à notre besoin de suivre un processus de transformation, car elle propose des groupes de matières premières (appelées dans FoodEx2 « Raw Primary Commodities », RPC) et de produits dérivés de plus en plus élaborés (appelés « RPC derivatives » ou « Composite food ») obtenus par les différents procédés ou traitements. Néanmoins, certains libellés ont été modifiés par souci de simplification pour désigner les concepts. L’ensemble des libellés de FoodEx2 a été traduit en français en deux temps : tout d’abord, une traduction initiale a été effectuée par l’Anses en 2016 sur la version de 2015 de FoodEx2 (cette traduction a été en grande partie réalisée par un prestataire de traduction et amendée ensuite par les équipes de l’Anses), puis une mise à jour a été réalisée par INRAE en 2023-2024 corrigeant certains termes et ajoutant les nouveaux termes apparus depuis 2016. Cette récente mise à jour a impliqué des chercheurs et scientifiques INRAE experts du domaine. Par ailleurs, les définitions issues de FoodEx2 ont également été légèrement modifiées afin de respecter les critères établis pour bien décrire les concepts (Clés & Aubin, 2021). Tous les codes correspondants aux groupes d’origine ont néanmoins été conservés, ce qui a permis de garder une correspondance stricte entre les groupes FoodEx2 et les classes de concepts de PO²/TransformON et facilite la maintenance des alignements.
La hiérarchie « Substances non alimentaires » a été créée dans PO²/TransformON pour regrouper toutes les substances utilisées ou produites au cours des processus de transformation ou de caractérisation, en tenant compte de leur rôle ou usage : produits et réactifs intervenant dans les réactions chimiques, enzymes, matériaux d’emballage, fluides frigorigènes, émissions polluantes, ressources énergétiques et déchets recyclables. Elle comporte 252 classes. Notons que la construction de la branche « déchets recyclables » s’est appuyée sur les référentiels existants tels que le Catalogue européen des déchets (EWC, https://www.eea.europa.eu/help/glossary/eea-glossary/european-waste-catalogue-1). En suivant la même logique que pour les branches « Food » et « Feed » de FoodEx2, à savoir le degré de transformation, nous avons créé des classes de « déchets ou résidus organiques primaires » (déchets de tissus animaux ou végétaux ou résidus de la production agricole), de « déchets ou résidus organiques secondaires » (résidus ou sous-produits des processus de transformation) et de « biodéchets finaux » (déchets organiques liquides ou solides, y compris les boues et les déchets liquides provenant du traitement des déchets) en tenant compte de la classification SINOE (https://www.sinoe.org) dont l’éditeur est l’Agence de la transition écologique (Ademe).
PO²/Process
Deux types de processus ont été définis dans l’ontologie PO²/TransformON.
- Les processus planifiés sont contrôlables, dans le sens où ils suivent des procédures ou plans d’expérience. Ils sont alignables avec la classe obi:planned process.
- Les processus biologiques ou physiologiques tels que les fermentations ou la digestion humaine sont des processus non planifiés qui sont des phénomènes observables (ils ne sont pas complètement contrôlables, on peut seulement les orienter en jouant sur des paramètres de conduite). Ils sont alignables avec la classe bfo:process.
La hiérarchie des process comporte 36 classes.
PO²/Step
Dans le modèle PO², les étapes (PO²/Step) sont les éléments qui composent un processus et qui peuvent être des changements d’état ou des phénomènes physiques, des étapes de processus biologiques ou physiologiques, des étapes de caractérisation ou des étapes de transformation. Ces quatre classes ou catégories ont été divisées en sous-classes plus fines spécialisant les étapes en fonction du type d’action ou de phénomène qu’elles représentent. Par exemple, les étapes de transformation ont été divisées en plusieurs niveaux en fonction de la nature de l’opération impliquée : biologique, chimique ou biochimique, et physique. La liste des étapes ou opérations a été établie à partir de la hiérarchie des procédés de FoodEx2 et des connaissances issues de la bibliographie ou provenant des experts. La hiérarchie des steps comporte 545 classes.
Partie « Observation » (PO²/Material, PO²/Method, PO²/Scale)
La partie « Observation » contient trois hiérarchies : PO²/Material, PO²/Method, PO²/Scale (Figure 7).
La partie « Observation » de PO²/TransformON comprend les concepts qui permettent de décrire les matériels, les méthodes et les échelles d’observation ou de transformation. Les concepts PO²/Material sont définis comme des systèmes (sosa:System), qui peuvent être composés de sous-systèmes et sont de deux natures différentes, soit un équipement de transformation (sosa:Actuator), soit un instrument de mesure (sosa:Sensor), qui peuvent être un appareil ou des agents humains (par exemple, un jury de dégustation). Les concepts de la hiérarchie PO²/Method représentent des procédures, des modes opératoires ou des protocoles analytiques (sosa:Procedure).
Au niveau d’une observation, on va pouvoir indiquer si les caractéristiques ou variables que l’on va chercher à observer ou quantifier portent sur un produit entrant ou sortant d’une étape ou sur le procédé lui-même. Les concepts spécialisant PO²/Scale permettent d’indiquer l’échelle de l’objet observé (échelle de mesure) ou à quelle échelle est réalisé le procédé (échelle de transformation). La hiérarchie des méthodes comporte 491 classes, celle des matériels 455 classes et celle des échelles de mesure et de transformation 20 classes. Une observation PO² permet de regrouper différents tableaux comportant les résultats de mesures ou de calculs.
Seuls les trois premiers niveaux de la hiérarchie sont montrés
Figure 7. Détail des hiérarchies PO²/Material, PO²/Method, PO²/Scale et PO²/Attribute
Partie « Résultat » (PO²/Attribute, unités de mesure et métadonnées de provenance)
La hiérarchie PO²/Attribute regroupe les caractéristiques observables (mesurables ou calculables) comme montré sur la Figure 7. La hiérarchie est divisée en quatre parties : 1) les résultats de calcul (caractéristiques dont les valeurs sont calculées à partir d’autres valeurs, comme des valeurs d’indice ou des scores), 2) les propriétés intrinsèques (caractéristiques inhérentes aux objets), 3) les attributs de mesure (caractéristiques obtenues directement à partir d’une mesure) ; et 4) les paramètres opératoires (caractéristiques permettant de contrôler les procédés de transformation ou de caractérisation). De nombreux attributs proviennent de FoodEx2, et la plupart a ensuite été créée en fonction des variables à décrire. La liste des paramètres de contrôle sera complétée pour d’autres types de techniques, selon les besoins.
Les attributs de mesure ont été organisés en sous-classes en fonction de leur nature : attributs biochimiques, biologiques, mensuration, perception sensorielle, attributs physico-chimiques, quantité, taux, ratios, mesures semi-empiriques, rendements.
Par ailleurs, une unité est associée aux valeurs quantitatives à l’aide du système UCUM. Des métadonnées sont également renseignées à l’aide du standard DCAT (https://www.w3.org/TR/vocab-dcat-3), par exemple pour indiquer les dates associées au jeu de données, le nom du projet, des descriptions sous forme textuelle ou des liens vers des fichiers externes. Des concepts de l’ontologie PROV-O (https://www.w3.org/TR/prov-o) sont réutilisés pour indiquer les organisations ou les agents (prov:Agent) en charge de réaliser les opérations de transformation ou les caractérisations (définies par ailleurs comme sous-classes de prov:Activity).
Mise en œuvre du FAIR-by-design à l’aide des outils développés
Pour mettre en œuvre le FAIR-by-design, il est nécessaire de concevoir la structuration des données dès le démarrage du projet de recherche, en se basant sur le modèle PO² et le vocabulaire TransformON présentés ci-dessus. Pour réaliser la structuration et la saisie des données, nous avons développé un logiciel, appelé PO² Manager.
Les Figures 8A-C présentent des captures d’écran constituant l’interface de saisie de ce logiciel.
La Figure 8A montre sur la partie gauche l’ensemble des itinéraires associés à un projet PO². Les itinéraires représentent des variations de compositions, de paramètres de contrôle des procédés ou d’enchaînement d’étapes dans un processus.
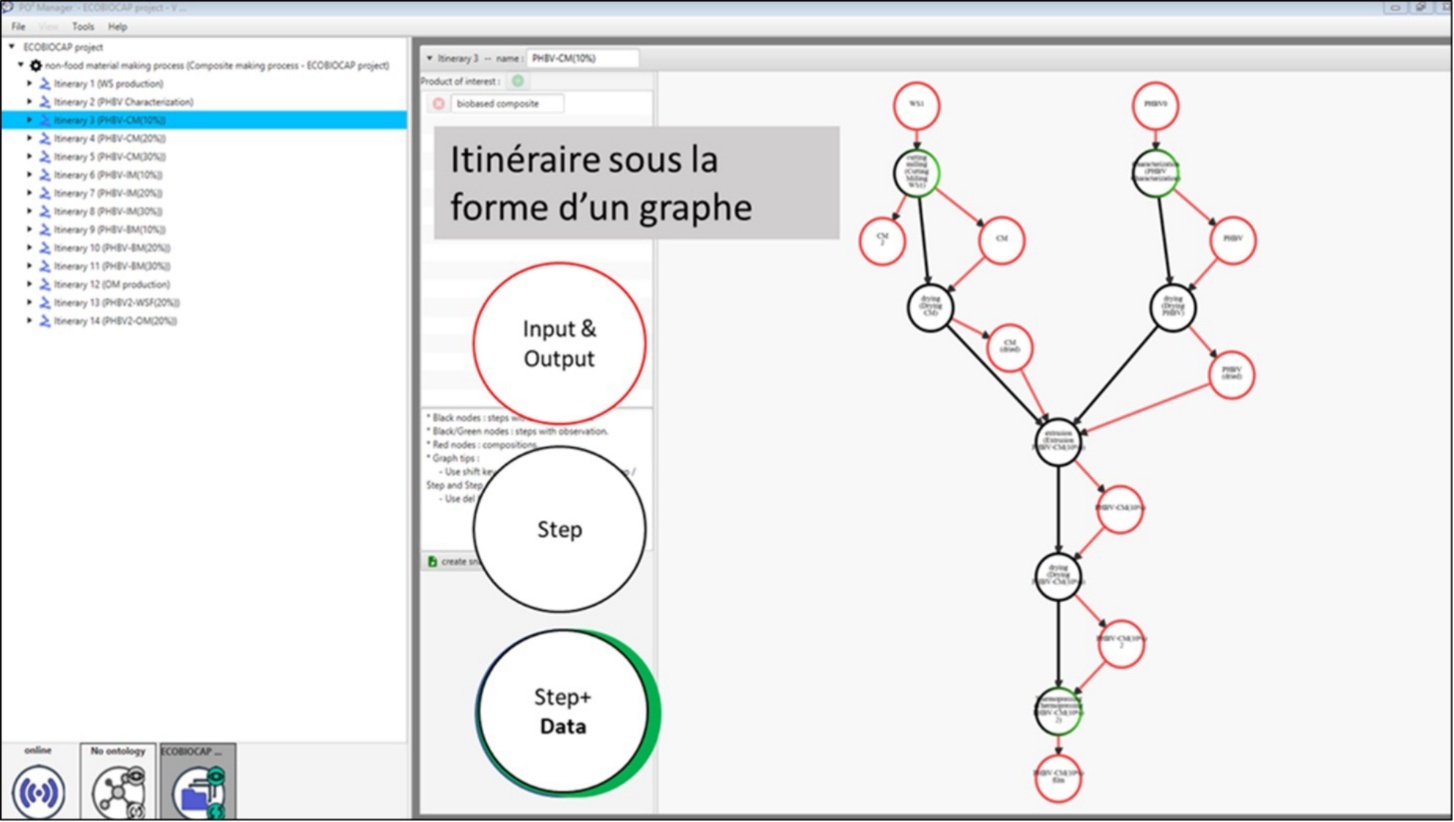
Figure 8A. Vue montrant la partie graphique de PO² Manager pour la gestion des itinéraires
La Figure 8A montre sur la partie droite un itinéraire matérialisé sous la forme d’un graphe avec une succession d’étapes (Step) représentées par des cercles noirs, et des composants ou compositions d’entrée ou de sortie (Input & Output) représentés par des cercles rouges. Les étapes contenant des observations (Step+Data) sont indiquées par un cerclage noir et vert. Les étapes sont reliées entre elles par des flèches noires qui représentent les enchaînements temporels. Le sens de la flèche rouge entre la composition et l’étape indique si la composition est entrante ou sortante. On peut représenter des itinéraires ayant des voies en parallèle (sur cet exemple on voit que deux branches d’opérations se rejoignent). Sur la partie gauche de l’interface, on voit la liste des itinéraires regroupés sous un « Process ». Un « Process » PO² regroupe également les réplicats d’itinéraires, c’est-à-dire des enchaînements de steps et d’observations réalisés dans les mêmes conditions, sur des matières premières (bioressources) similaires afin d’observer la variabilité inhérente au processus ou à l’échantillonnage. Un réplicat est une nouvelle réalisation de l’expérimentation dans les conditions les plus identiques possibles en fonction d’un plan d’expérience donné correspondant à un ou plusieurs facteurs testés. Les répétitions de mesure individuelles réalisées sur un échantillon donné au cours de la même expérimentation sont quant à elles représentées dans les tableaux d’observation.
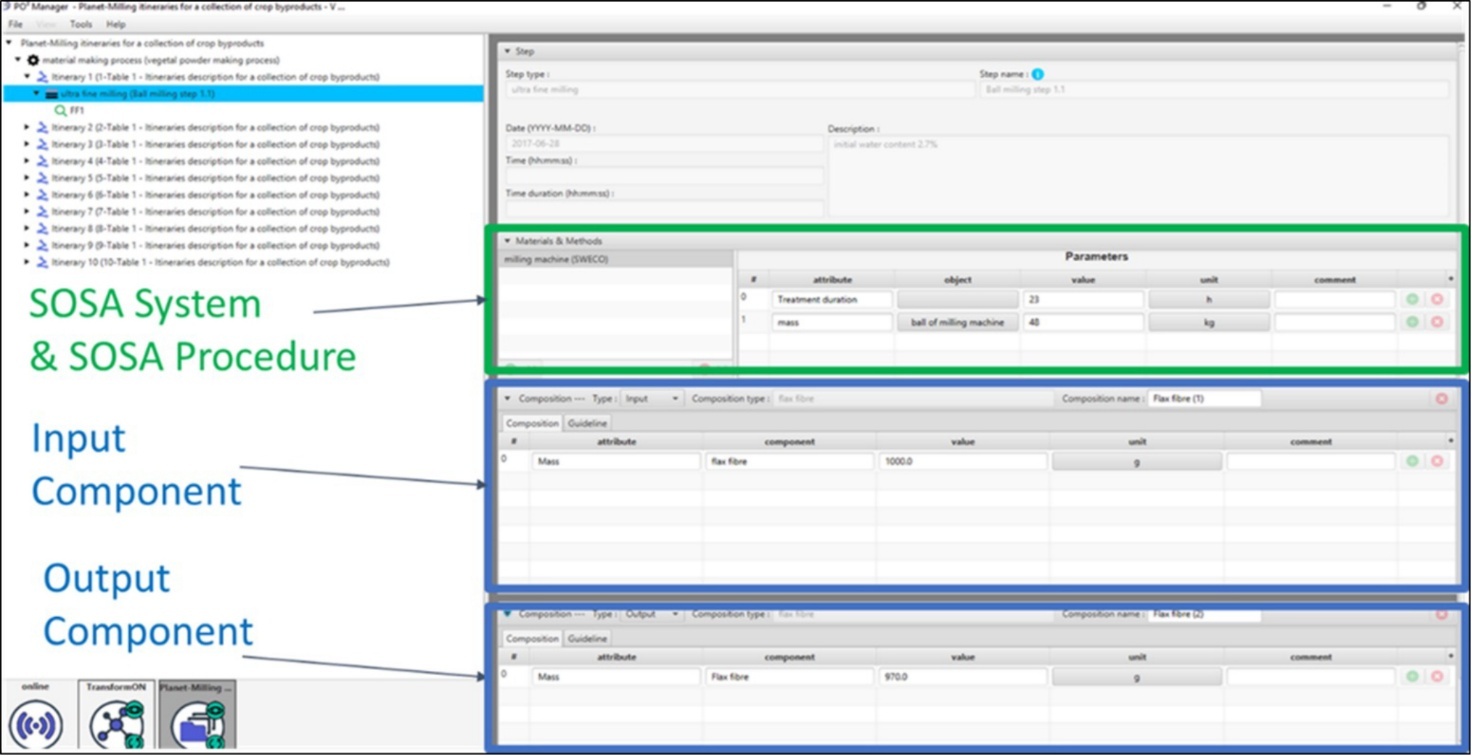
Figure 8B. Vue de l’écran de saisie d’un step
L’expérimentateur remplit la zone en haut à droite qui contient les informations générales de l’étape (Figure 8B), qui sont destinées à indiquer la provenance des données et les dates pour assurer la traçabilité. Puis, toujours à droite, une zone (encadré vert) pour indiquer les matériels (sosa:System) et les méthodes (sosa:Procedure) et deux zones contenant le détail des compositions d’entrée et de sortie de cette étape (encadrés bleus).
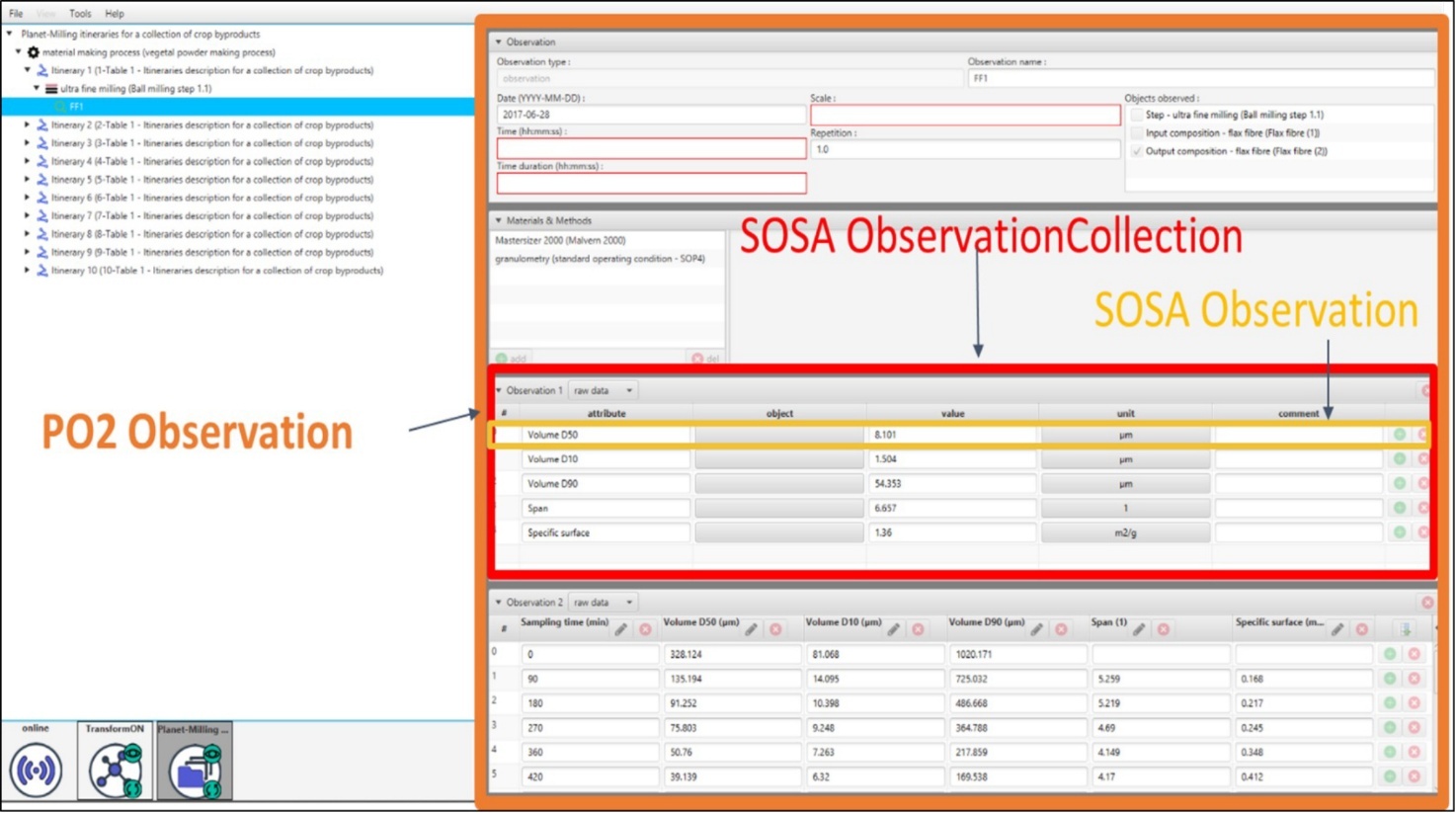
Figure 8C. Vue de l’écran de saisie des observations
La Figure 8C montre les zones permettant la saisie des données dans des observations PO² (encadré orange). On peut ajouter les informations relatives à l’observation (dates, opérateurs, objet observé, description), indiquer les matériels et méthodes utilisés et insérer des tableaux contenant les collections d’observations SOSA (encadré rouge). Les matériels et méthodes sont décrits à l’aide de l’ontologie au niveau du projet. Une observation PO² peut contenir plusieurs tableaux de données, ce qui implique qu’ils ont été réalisés à l’aide d’un même matériel. Un tableau représente une collection d’observations (sosa:ObservationCollection). Les résultats contenus dans les tableaux peuvent être des mesures brutes ou dériver de calculs. Les tableaux peuvent contenir des observations simples sous la forme de lignes ou être des tableaux complexes contenant une colonne par variable. Chaque ligne ou colonne représente une sosa:Observation (encadré jaune). Les répétitions de mesure sont représentées sous la forme de tableaux complexes.
Afin de standardiser la conception des itinéraires, des modèles de fichier Excel (templates) associés aux steps et aux observations ont été définis avec le vocabulaire standardisé PO²/TransformON. Ces fichiers peuvent être importés directement via l’outil PO² Manager.
Toutes les caractéristiques et les objets d’intérêt sur lesquels portent les attributs sont typés à l’aide de l’ontologie de domaine, ce qui permet la sémantisation, c’est-à-dire la création du graphe RDF avec un vocabulaire contrôlé.
Une fois les données sémantisées sous la forme de graphes, elles sont publiées dans la base de données graphes au format RDF nommée PO²-BaGaTel, et peuvent être interrogées à l’aide du langage SPARQL, un standard du Web sémantique. Une application web appelée Simple PO²Query (SPO²Q) a également été développée pour permettre aux utilisateurs ne connaissant pas le langage SPARQL d’interroger la base, à travers une interface permettant de les guider pas à pas pour générer les requêtes. Les tableaux de données issus des requêtes sont exportables sous forme structurée (format TSV ou JSON) tandis que les requêtes peuvent être enregistrées pour permettre leur réutilisation, ou modifiées à l’aide d’un éditeur en mode avancé. Les données peuvent ensuite aussi être déposées dans des entrepôts comme Recherche Data Gouv (reposant sur la solution Dataverse) ou d’autres entrepôts thématiques, en accord avec les principes FAIR.
La Figure 9 présente une vue d’ensemble de la démarche de structuration à l’aide de l’écosystème logiciel développé autour de PO²/TransformON : PO² Manager et SPO²Q (Dervaux et al., 2023 ; Dervaux et al., 2024a ; Dervaux et al., 2024b). Les logiciels ont fait l’objet d’un dépôt dans l’entrepôt Software Heritage (https://www.softwareheritage.org) et dans HAL INRAE (https://hal.inrae.fr).
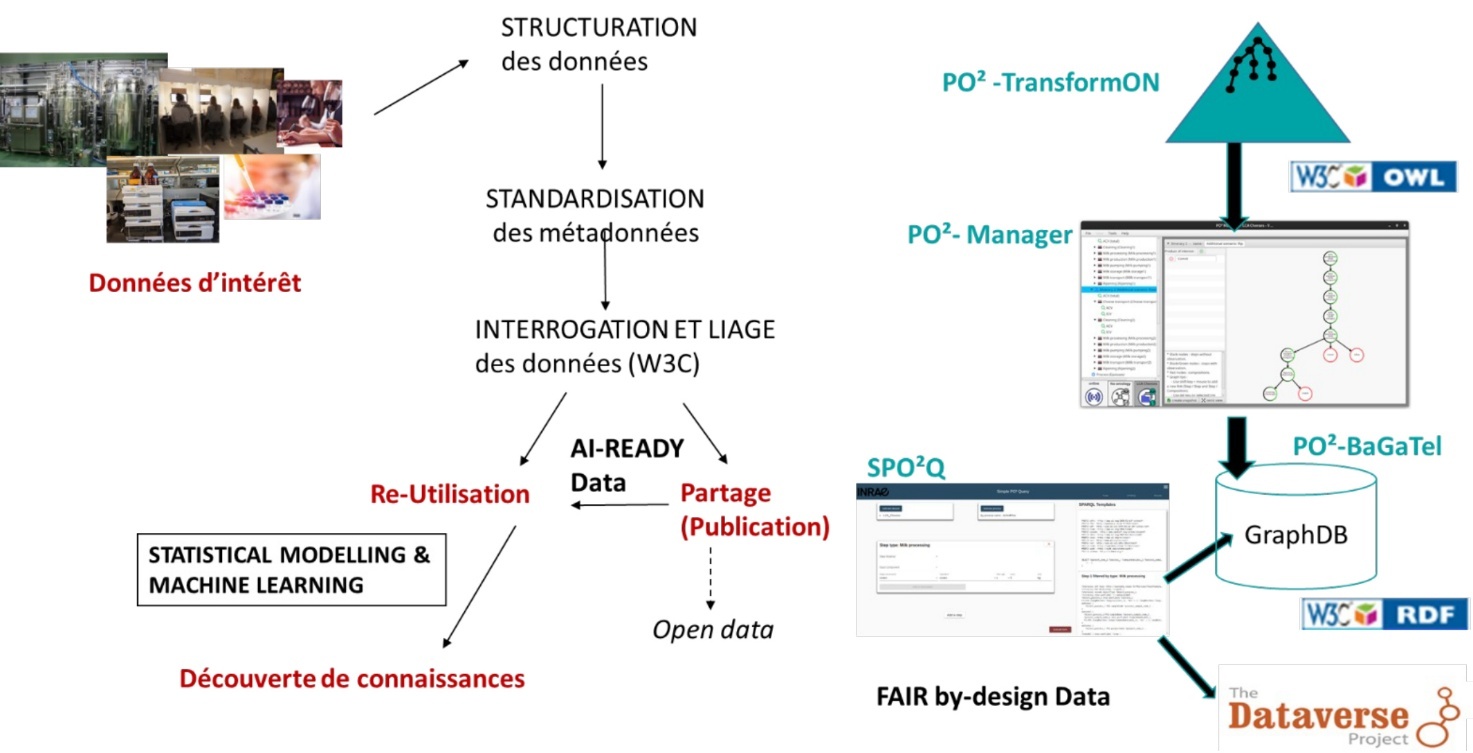
Figure 9. L’écosystème d’outils basés sur le Web sémantique et PO²/TransformON
L’ontologie PO²/TransformON et les outils logiciels PO² Manager et SPO²Q sont développés en interne. La base de données graphe (aussi appelée triple store RDF) est gérée avec la solution commerciale GraphDB (https://graphdb.ontotext.com).
Exemples d’application sur les domaines alimentaire et non alimentaire
Nous présentons ici deux exemples d’application sur les domaines alimentaire et non alimentaire illustrant le couplage de modèles et l’intégration de données hétérogènes portant sur les multiples dimensions des aliments ou des emballages biosourcés (Figures 10A-B).
Écoconception de procédés alimentaires et d’aliments sains, durables et sensoriellement satisfaisants
Dans les travaux sur l’écoconception, la place des données et des connaissances est centrale, en particulier si on souhaite mobiliser l’Analyse du cycle de vie ou ACV (ISO 14040 : 2006
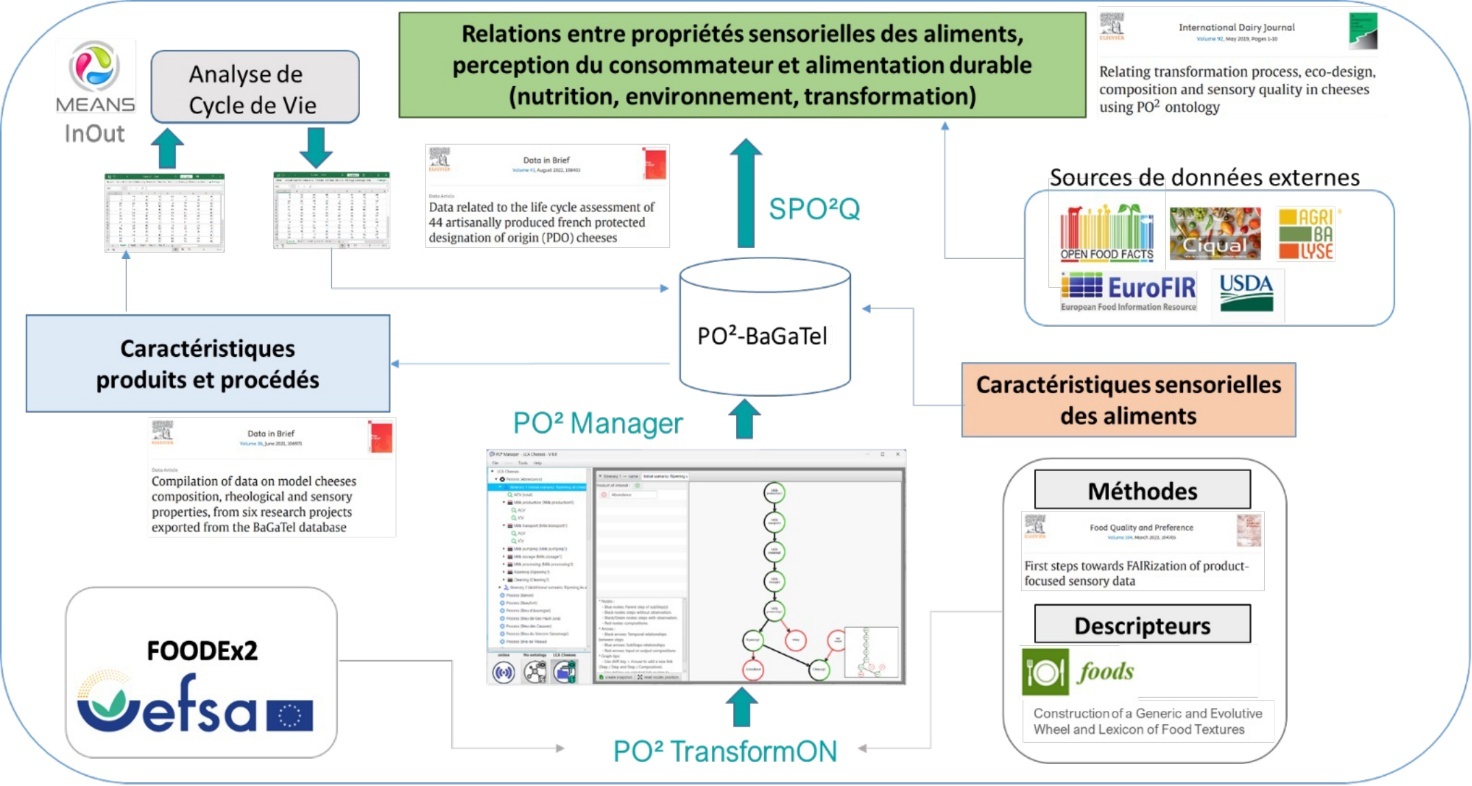
Figure 10A. Utilisation de PO²/TransformON pour la mise en relation des caractéristiques produits et procédés et la conception d’aliments sains, durables et sensoriellement satisfaisants
Dans une première approche, la base de données PO²-BaGaTel a été interrogée pour répondre à des questions portant sur l’écoconception de la transformation du fromage à pâte dure, en relation avec la composition, la qualité sensorielle et les propriétés rhéologiques (Pénicaud et al., 2019). Des données de qualité des fromages à pâte pressée existaient dans la base (Allard et al., 2018), que nous voulions compléter d’une estimation des impacts environnementaux générés par la production du fromage. Des requêtes dans la base de données ont permis de quantifier les quantités d’énergies et de matières (matières premières, produits de nettoyage…) nécessaires à l’inventaire de cycle de vie. L’ensemble des données obtenues peut ensuite être utilisé pour réaliser une ACV (Cortesi et al., 2022a ; Cortesi et al., 2022b). Les calculs de l’ACV se font de façon habituelle, indépendamment de la base PO²-BaGaTel, à l’aide d’un logiciel d’ACV (e. g. SimaPro, https://network.simapro.com/evea). Les résultats de l’ACV obtenus sous format tableur peuvent être intégrés à la base PO²-BaGaTel, en y associant l’échantillon étudié et toutes les métadonnées et hypothèses qui y sont liées. Ainsi, pour cet échantillon, nous disposons maintenant de données de composition, sensorielles, rhéologiques et d’impacts environnementaux. Le vocabulaire et la structure fournis par l’ontologie PO² ont permis de combiner et d’intégrer dans PO²-BaGaTel des données provenant de différents projets, donnant des réponses pertinentes à différentes questions, et prouvant ainsi son adéquation en tant qu’outil de soutien à l’évaluation multicritères des systèmes alimentaires.
Pour aller plus loin dans l’intégration de l’ACV et de PO², un travail a été conduit pour rendre interopérables PO²-BaGaTel et MEANS (Auberger et al., 2022). La plateforme MEANS héberge le logiciel MEANS-InOut, une application web conviviale qui aide les utilisateurs à appliquer l’ACV en construisant des inventaires du cycle de vie (ICV) à partir de données qui décrivent le système étudié. Dans MEANS-InOut, pour représenter un système de production/transformation des aliments et des bioproduits, les utilisateurs commencent par représenter le diagramme des flux. Un cadre générique a été développé pour décrire les étapes du procédé et guider la collecte de données : flux d’ingrédients et de produits, énergie, eau, pratiques de refroidissement, emballage, équipement, consommables, détergents, traitement des déchets ou de l’eau, émissions de polluants et transport (type et distance). Le vocabulaire de référence PO²/TransformON est utilisé pour entrer les noms des ingrédients, des produits, des procédés et des étapes, à l’aide de menus déroulants. En fonction de leur objectif, les utilisateurs peuvent ensuite exporter un ICV de procédé ou un ICV de produits et co-produits, et pour ces derniers, plusieurs méthodes d’allocation sont possibles : massique sur une base sèche ou humide, volumique, protéique, lipidique, énergétique ou économique. La création par MEANS-InOut de ces ICV de procédés ou de produits est basée sur une correspondance entre les flux consommés et émis par le procédé (ingrédients, énergie, machines, traitement des déchets...) et les ICV issus des bases de données Ecoinvent (https://ecoinvent.org) et Agribalyse (https://agribalyse.ademe.fr). Les fichiers d’ICV générés avec MEANS-InOut sont prêts à être importés dans un logiciel d’ACV, ce qui est nécessaire pour calculer les indicateurs d’impacts environnementaux. La possibilité d’interroger PO²-BaGaTel à partir de MEANS-InOut est prévue dans les développements ultérieurs, et la possibilité d’exporter les données d’ACV de MEANS-InOut vers la base de données PO²-BaGaTel permettra de les connecter à d’autres dimensions des produits alimentaires et de réaliser des évaluations multicritères appropriées.
Par ailleurs, un travail de mise en relation de données relatives aux procédés de fabrication, écoconception, composition et qualité des fromages a été initié à l’aide d’une première version de l’ontologie PO² (Guichard et al., 2021a ; Guichard et al., 2021b). Ce travail a permis de mettre en relation des données hétérogènes issues de trois projets collaboratifs et de treize publications scientifiques portant sur les paramètres de fabrication et les spécificités de certains fromages (composition, propriétés rhéologiques, propriétés sensorielles, activité masticatoire, libération du sodium et libération d’arômes). Il a ainsi été possible de mettre en évidence certaines relations entre les données sensorielles et les données de rhéologie de fromages à pâte pressée. Grâce à l’interrogation de la base PO²-BaGaTel, il a également été possible d’estimer les données manquantes en se basant sur des caractéristiques similaires ou comparables entre les échantillons. Par la suite, la base de données relationnelle a été transformée en base de données au format graphe pour pouvoir suivre l’évolution du modèle PO². Un travail d’identification et de structuration portant sur les descripteurs sensoriels (goût, odeur ou arôme, texture ou aspect) et sur les méthodes d’analyse sensorielle a ensuite été réalisé afin de disposer d’un vocabulaire partagé pour décrire les données relatives aux caractérisations organoleptiques des aliments (Bondu et al., 2022 ; Visalli et al., 2022). Enfin, une solution a été développée pour permettre d’aller interroger des sources externes comme les bases de données de compositions nutritionnelles établies par les agences comme l’Anses ou l’USDA (Buche et al., 2021). Ce travail sera étendu à d’autres sources comme la base de données Open Food Facts (https://fr.openfoodfacts.org) pour disposer des compositions et des listes d’ingrédients d’aliments du commerce.
Aide à la sélection de biomasse pour la conception d’emballages composites
Le développement de biocomposites via l’incorporation de fibres ligno-cellulosiques naturelles dans une matrice polymère biosourcée et biodégradable prend une importance croissante comme alternative possible aux plastiques conventionnels. Parmi les biopolymères prometteurs, le poly(3-hydroxybutyrate-co-3-hydroxyvalérate), ou PHBV, est un polyester bactérien qui présente l’avantage d’être biodégradable dans tous les environnements et de pouvoir être synthétisé à partir de tous types de résidus carbonés. L’incorporation de charges ligno-cellulosiques à faible coût, obtenues soit à partir de l’industrie de la pâte à papier, soit par fractionnement à sec de résidus ligno-cellulosiques (par exemple, sarments de vigne, marcs d’olive, paille de blé, déchets verts de parcs et jardins urbains, coques de pistaches…) permet de réduire le coût global des matériaux et leur impact environnemental, tout en modulant leurs performances. Dans ce contexte, l’ontologie PO²/TransformON a rendu possible la réutilisation de données expérimentales issues de différentes sources produites dans plusieurs projets nationaux ou européens pour aider à la formulation d’emballages alimentaires biocomposites (Figure 10B).
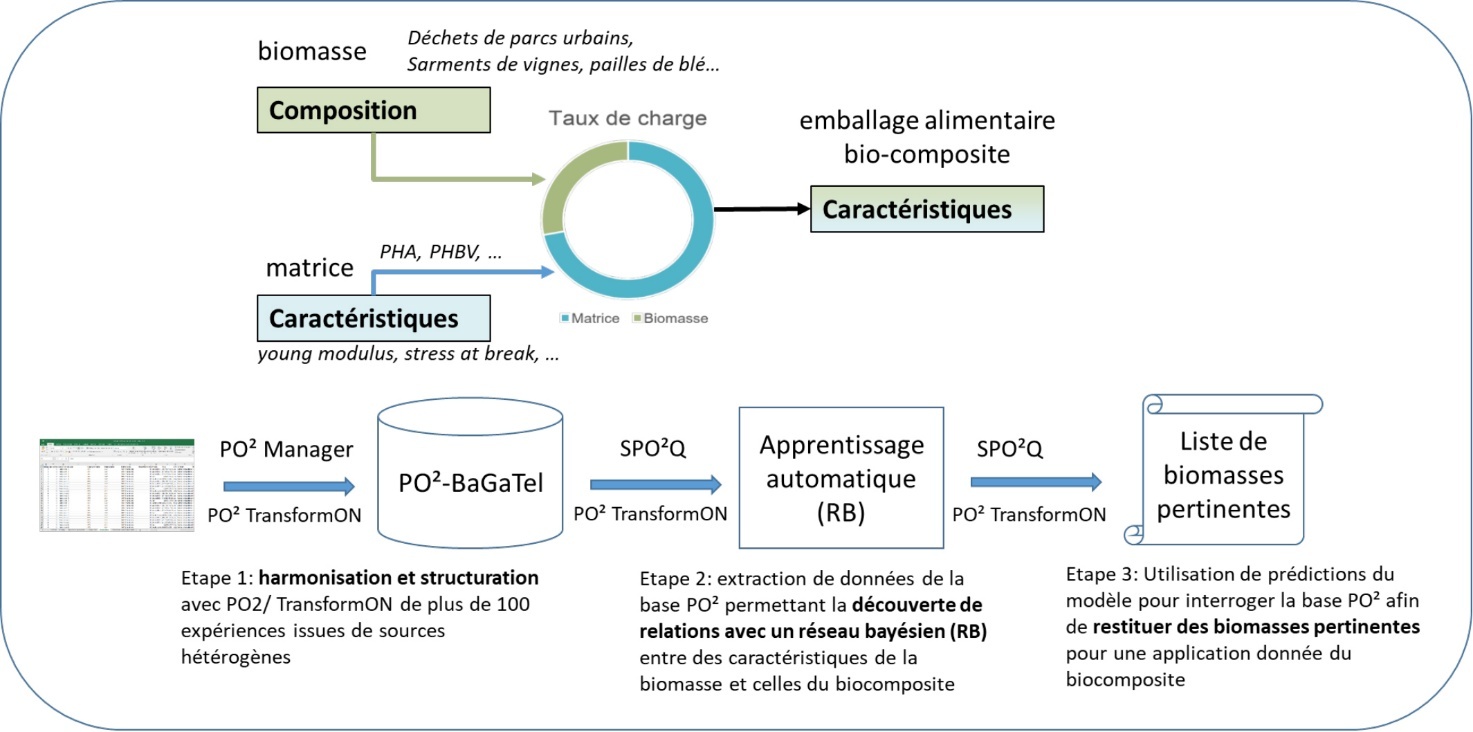
Figure 10B. Utilisation de PO²/TransformON pour une aide à la sélection de biomasse pour une application emballage donnée (RB : réseaux bayésiens)
Dans une première étape, l’harmonisation en termes de vocabulaire et de structuration des données provenant de cinq projets nationaux ou européens et issues de plus de 100 expériences a été réalisée avec l’ontologie PO²/TransformON, et les données ont été intégrées dans la base PO²-BaGaTel (Fabre et al., 2020). Ce travail d’harmonisation et de structuration a permis dans une seconde étape de réaliser une extraction des données de la base avec l’outil SPO²Q et la découverte de relations entre d’une part, la composition de la biomasse et d’autre part, les caractéristiques mécaniques et de perméabilité des emballages biocomposites, grâce à l’utilisation de modèles d’apprentissage automatique (Münch et al., 2022a ; Münch et al., 2022b). Enfin, le modèle appris a permis dans une troisième étape de prédire automatiquement les profils biochimiques des poudres végétales bien adaptées à la production d’emballages biocomposites ayant des caractéristiques mécaniques et de perméabilité ciblées. Une nouvelle interrogation avec SPO²Q d’autres sources de données enregistrées dans la base PO² contenant un catalogue de biomasses caractérisées biochimiquement a permis l’identification de biomasses adaptées au besoin (Münch et al., 2022c). Le couplage structuration des données au format PO²/TransformON-apprentissage automatique a permis de découvrir des relations à l’échelle d’un enchaînement d’étapes, ce qui n’avait jamais été réalisé avec de modèles déterministes. D’autre part, la structuration détaillée des itinéraires de transformation/caractérisation avec l’ontologie PO²/TransformON est un investissement sur le long terme qui permet d’une part la reproduction des expérimentations et d’autre part d’enrichir progressivement la base de connaissances pour traiter de nouvelles questions de recherche. Une perspective actuelle est d’aider à l’écoconception de nouveaux itinéraires de transformation de biomasse pour une application biocomposites donnée en prenant en considération non seulement les caractéristiques fonctionnelles des matériaux désirés mais également l’impact environnemental de la production de ceux-ci.
Conclusion et perspectives
L’ensemble des jeux de données structurés et annotés à l’aide de l’ontologie PO²/TransformON est interrogeable dans la base de données graphe et les jeux de données assemblés à l’aide des requêtes peuvent ensuite être déposés dans l’espace institutionnel Data INRAE au sein de l’entrepôt Recherche Data Gouv (https://entrepot.recherche.data.gouv.fr/dataverse/bagatel). L’ontologie de domaine PO²/TransformON fournit un vocabulaire conçu pour répondre aux besoins spécifiques de l’ingénierie des aliments, des bioproduits et des résidus ou déchets issus de biomasse. PO²/TransformON permet de couvrir l’ensemble du système de transformation de la biomasse en produits alimentaires ou non alimentaires en intégrant les co-produits ou résidus. Il s’agit de la première étape qui permettrait de mettre en relation la production, la transformation et la consommation dans le cadre d’une approche plus transversale.
L’ontologie constitue un système de référence fournissant des identifiants de ressources uniques sur le Web pour le vocabulaire harmonisé en anglais et en français et aligné avec FoodEx2. L’ontologie permet ainsi de capitaliser les données expérimentales, de les contextualiser en les documentant et de prendre en compte la connaissance experte. Le travail de définition et de partage d’un référentiel commun permet de favoriser les échanges interdisciplinaires, mais il est nécessaire de sensibiliser et former davantage d’utilisateurs et de systématiser le travail de FAIRification des données pour de nouveaux projets dès leur démarrage (FAIR-by-design). Nous avons montré deux exemples d’application mais de nombreux autres cas d’étude portent soit sur les performances de procédés, soit sur les caractéristiques finales et propriétés d’usage de produits alimentaires ou non alimentaires. Un des travaux en cours est de compléter le vocabulaire TransformON par une extension dédiée aux bioprocédés, c’est-à-dire les procédés impliquant des agents biologiques comme les fermentations (projet FermentON). Sur le périmètre alimentaire, nous développons une approche visant à développer une ontologie complémentaire à TransformON (Weber et al., 2025), axée sur les sciences du consommateur (module ConsomON). La suite du travail consiste à créer les définitions des concepts encore non définis et à aligner la partie terminologie de l’ontologie avec le Thésaurus INRAE pour l’interopérabilité avec les systèmes d’information institutionnels. Des alignements avec d’autres ontologies (notamment l’ontologie FoodOn) seront recherchés pour constituer un réseau d’ontologies interconnectées et faciliter ainsi l’intégration d’autres jeux de données. Le vocabulaire TransformON spécialisé sur le domaine de la bioéconomie et de l’alimentation peut aussi être mobilisé en tant que référentiel pivot pour permettre à des systèmes de communiquer entre eux, à l’instar de l’interfaçage avec la plateforme MEANS pour la réalisation des ACV, ou pour d’autres applications impliquant plusieurs agents ou composants logiciels. Des développements dans ce sens sont en cours dans le cadre du projet ICAM (Buche et al., 2024) visant à prototyper une plateforme numérique intégrative de connaissances dans une approche de science ouverte, sur un périmètre donné de bioraffinerie, la production et la valorisation de poudres végétales. Cette plateforme permettra aux acteurs s’intéressant à la bioéconomie d’avoir accès à des données expérimentales, de trouver des réponses à des questions scientifiques et d’obtenir des recommandations argumentées dans la mise en œuvre d’une bioraffinerie durable. Enfin, l’ontologie peut être couplée à d’autres modèles pour l’apprentissage automatique et enrichir des modèles d’IA génériques pré-entraînés avec des données expertes, ce qui ouvre de nouvelles perspectives pour répondre aux enjeux liés à l’alimentation, la santé et la bioéconomie.
Accessibilité
L’ontologie de domaine PO²/TransformON est disponible avec une licence Etalab 2.0 (équivalent Creative Commons CC0) dans l’entrepôt Recherche Data Gouv dans la collection Data INRAE : https://doi.org/10.57745/DWX7W6
Elle est également déposée dans AgroPortal : https://agroportal.lirmm.fr/ontologies/TRANSFORMON
Remerciements
Le projet AIC TransformON a été financé par le département INRAE TRANSFORM sur la période 2021-2022. Un financement conjoint MICA-TRANSFORM a permis son extension aux bioprocédés (projet AIC FermentON) sur la période 2023-2025.
Nous remercions l’ensemble des contributeurs de l’ontologie appartenant aux unités du département TRANSFORM ou MICA participant aux projets TransformON et FermentON (SAYFOOD, STLO, SPO, UEPR, LBE, PROSE, IATE, TBI, BIA, CSGA et QUAPA) : Marie Debacq, Hélène Falentin, Anne Thierry, Florence Valence-Bertel, Valérie Gagnaire, Gaëlle Tanguy, Émeline Goussé, Geneviève Gesan-Guiziou, Mélanie Münch, Marc Perez, Evelyne Aguera, Florence Jacques, Éric Latrille, Élie Le Quemener, Virginie Rossard, Émilie Fernandez, Dimitri Martel, Ariane Bize, Estelle Grousseau, Jules Fievet, Thibault Magnac, Hélène Angellier-Coussy, Claire Mayer, Valérie Guillard, Reine Barbar, Adrien Réau, Alrick Oudot, Emmanuele Piaud, Leslie Lhomond, Luc Menut, David-Camilo Corrales-Munoz, Juliane Rolland, Véronique Solé-Jamault, Alice Kermarrec, Catherine Garnier, Alain Riaublanc, Adeline Boire, Hanitra Rabesona, Valérie Beaumal, Lucie Birault, Audrey Geairon, Camille Jonchère, Adeline Dourson, Kayah Esnault, Elisabeth Guichard, Christian Salles, Chloé Lombardi, Caroline Bondu, Thomas Delompré, Carole Tournier, Anne Tromelin, Chantal Septier, Christophe Martin, Claire Follot, Hélène Brignot, Hélène Labouré, Noëlle Béno, Thierry Thomas-Danguin, Géraldine Lucchi, Isabelle Andriot, Amidou Traoré, Sylvie Clerjeon, ainsi qu’Arnaud Charleroy de l’unité MISTEA (département MATHNUM) pour le travail en lien avec le continuum vigne-vin et Cédric Baudrit, Christophe Fernandez de l’unité I2M Bordeaux pour leur collaboration pour les couplages de modèles et la représentation des connaissances.
Nos remerciements vont aussi à Sonia Bravo et Anne-Sophie Bage, membres de la DipSO, pour leur relecture et leur support pour la rédaction.
Notes
- 1. Thésaurus INRAE. https://consultation.vocabulaires-ouverts.inrae.fr/thesaurus-inrae/fr/
- 2. World Health Organization : WHO (2023). Une seule santé (consulté le 1er avril 2025). https://www.who.int/fr/news-room/fact-sheets/detail/one-health
- 3. Objectifs de développement durable. Nations Unies (consulté le 1er avril 2025). https://www.un.org/sustainabledevelopment/fr/objectifs-de-developpement-durable/
- 4. Produire des données FAIR. La Science Ouverte À INRAE (consulté le 2 avril 2025). https://science-ouverte.inrae.fr/fr/le-numerique-pour-la-science-et-les-donnees-scientifiques/produire-des-donnees-fair
- 5. European commission, 2000/532/EC. Commission Decision of 3 May 2000 replacing Decision 94/3/EC establishing a list of wastes pursuant to Article 1(a) of Council Directive 75/442/EEC on waste and Council Decision 94/904/EC
- 6. ISO 14040 : 2006. ISO. https://www.iso.org/standard/37456.html
Références
- Allard, T., Guichard, E., Perret, B., Guillemin, H., & Pénicaud, C. (2018). Mise en relation de données relatives aux procédés de fabrication, écoconception, composition et qualité des produits laitiers à l’aide d’une base de données guidée par une ontologie. NOV’AE (Cahier des techniques), Spécial 02, 1-11. https://revue-novae.fr/article/view/8802
- Andrés-Hernández, L., Blumberg, K., Walls, R. L., Dooley, D., Mauleon, R., Lange, M., Weber, M., Chan, L., Malik, A., Møller, A., Ireland, J., Segovia, L., Zhang, X., Burton-Freeman, B., Magelli, P., Schriever, A., Forester, S. M., Liu, L., & King, G. J. (2022). Establishing a Common Nutritional Vocabulary - From Food Production to Diet. Frontiers In Nutrition, 9, 928837. https://doi.org/10.3389/fnut.2022.928837
- Auberger, J., Gésan-Guiziou, G., Malnoë, C., Rostain, G., Weber, M., Guillemin, H., Dervaux, S., & Pénicaud, C. (2022). Adapting MEANS-InOut LCA software to food engineering, in relation to the PO2 food ontology and PO2-BaGaTel food engineering database. 13th International Conference on Life Cycle Assessment of Food 2022 (LCA Foods 2022), Octobre 2022, Lima, Perou. https://hal.inrae.fr/hal-03837121v1
- Aubin, S., Bernard, É., Bravo, S., Cadiou, C., Girard, A., & Weber, M. (2025). Révéler et valoriser vos données avec le thésaurus INRAE. HAL (le Centre Pour la Communication Scientifique Directe). https://doi.org/10.17180/novae-2024-no-art03
- Bondu, C., Salles, C., Weber, M., Guichard, E., & Visalli, M. (2022). Construction of a Generic and Evolutive Wheel and Lexicon of Food Textures. Foods, 11(19), 3097. https://doi.org/10.3390/foods11193097
- Buche, P., Cufi, J., Dervaux, S., Dibie, J., Ibanescu, L., Oudot, A., & Weber, M. (2021). How to Manage Incompleteness of Nutritional Food Sources ? International Journal Of Agricultural And Environmental Information Systems, 12(4), 1 26. https://doi.org/10.4018/ijaeis.20211001.oa4
- Buche, P., Reau, A., Oudot, A., Sablayrolles, C., Baudrit, C., Fernandez, C., Mayer-Laigle, C., Vialle, C., Belaud, J., Cufi, J., Lhomond, L., Weber, M., Ibanescu, L., Ouaret, R., & Dervaux, S. (2024). Projet ICAM (2024-26) : Ingénierie des Connaissances et Analyse Multidimensionnelle de la Durabilité pour valoriser les données, les connaissances et l’expertise scientifique des plateformes de bioraffinerie. Séminaire chercheurs Institut Carnod 3BCAR 2024, Janvier 2024, Montpellier, France. https://hal.science/hal-04475584
- Clés, Q., & Aubin, S. (2021). Rédiger une définition, quelques clés. Institut national de la recherche pour l’agriculture, l’alimentation et l’environnement. HAL (le Centre Pour la Communication Scientifique Directe). https://doi.org/10.15454/93xg-p646
- Cortesi, A., Dijoux, L., Bris, G. Y., & Pénicaud, C. (2022a). Data related to the life cycle assessment of 44 artisanally produced french protected designation of origin (PDO) cheeses. Data In Brief, 43, 108403. https://doi.org/10.1016/j.dib.2022.108403
- Cortesi, A., Dijoux, L., Bris, G. Y., & Pénicaud, C. (2022b). Explaining the Differences between the Environmental Impacts of 44 French Artisanal Cheeses. Sustainability, 14(15), 9484. https://doi.org/10.3390/su14159484
- Dervaux, S., Cufi, J., Guillemin, H., Weber, M., Ibanescu, L., Oudot, A., & Buche, P. (2024a). Simple PO2 Query (SPO2Q), a querying tool to retrieve biomass transformation and characterization itineraries using the Process and Observation (PO2) Ontology, 2024. https://hal.science/hal-04501660
- Dervaux, S., Guillemin, H., Cufi, J., Buche, P., Weber, M., & Ibanescu, L. (2023). PO2 Manager, an annotation tool to edit biomass transformation and characterization itineraries using the Process and Observation (PO2) Ontology. https://hal.science/hal-04313202
- Dervaux, S., Guillemin, H., Cufi, J., Buche, P., Weber, M., Ibanescu, L., & Oudot, A. (2024b). PO2 Engine, an application programming interface associated with the Process and Observation (PO2) software ecosystem, 2024. https://hal.science/hal-04387669
- Dooley, D., Andres-Hernandez, L., Bordea, G., Carmody, L., Cavalieri, D., Chan, L., Castellano-Escuder, P., Lachat, C., Mougin, F., Vitali, F., Yang, C., Weber, M., McGinty, H. K., & Lange, M. (2024). OBO Foundry food ontology interconnectivity. Semantic Web, 15(4), 1239 1258. https://doi.org/10.3233/sw-233458
- Dooley, D. M., Griffiths, E. J., Gosal, G. S., Buttigieg, P. L., Hoehndorf, R., Lange, M. C., Schriml, L. M., Brinkman, F. S. L., & Hsiao, W. W. L. (2018). FoodOn : a harmonized food ontology to increase global food traceability, quality control and data integration. Npj Science Of Food, 2(1), 23. https://doi.org/10.1038/s41538-018-0032-6
- Fabre, C., Buche, P., Rouau, X., & Mayer-Laigle, C. (2020). Milling itineraries dataset for a collection of crop and wood by-products and granulometric properties of the resulting powders. Data In Brief, 33, 106430. https://doi.org/10.1016/j.dib.2020.106430
- Guichard, E., Thomas-Danguin, T., Buchin, S., Perret, B., Guillemin, H., Pénicaud, C., & Salles, C. (2021a). Compilation of data on model cheeses composition, rheological and sensory properties, from six research projects exported from the BaGaTel database. Data In Brief, 36, 106971. https://doi.org/10.1016/j.dib.2021.106971
- Guichard, E., Thomas-Danguin, T., Buchin, S., Perret, B., Guillemin, H., Pénicaud, C., & Salles, C. (2021b). Relationships between cheese composition, rheological and sensory properties highlighted using the BaGaTel database. International Dairy Journal, 118, 105039. https://doi.org/10.1016/j.idairyj.2021.105039
- Ibanescu, L., Dibie-Barthelemy, J., Dervaux, S., Guichard, E., & Raad, J. (2016). PO2. A Process and Observation Ontology in Food Science. Application to Dairy Gels. MTSR. 155-165. https://doi.org/10.1007/978-3-319-49157-8_13
- Münch, M., Buche, P., Dervaux, S., Dibie, J., Ibanescu, L., Manfredotti, C., Wuillemin, P., & Angellier-Coussy, H. (2022a). Combining ontology and probabilistic models for the design of bio-based product transformation processes. Expert Systems With Applications, 203, 117406. https://doi.org/10.1016/j.eswa.2022.117406
- Münch, M., Buche, P., Manfredotti, C., Wuillemin, P., & Angellier-Coussy, H. (2022b). Une approche d’ingénierie inverse combinant ontologies et modèles relationnels probabilistes : application aux emballages bio-composites. IC@PFIA 2022 - Plate-Forme Intelligence Artificielle, Juin 2022, Saint-Etienne, France. https://hal.science/hal-03682416
- Münch, M., Buche, P., Manfredotti, C., & Wuillemin, P. H. (2022c). Formalising contextual expert knowledge for causal discovery in linked knowledge graphs about transformation processes : application to processing of bio-composites for food packaging. International Journal Of Metadata Semantics And Ontologies, 16(1), 1 15. https://doi.org/10.1504/ijmso.2022.131129
- Pénicaud, C., Ibanescu, L., Allard, T., Fonseca, F., Dervaux, S., Perret, B., Guillemin, H., Buchin, S., Salles, C., Dibie, J., & Guichard, E. (2019). Relating transformation process, eco-design, composition and sensory quality in cheeses using PO2 ontology. International Dairy Journal, 92, 1 10. https://doi.org/10.1016/j.idairyj.2019.01.003
- Poveda-Villalón, M., Fernández-Izquierdo, A., Fernández-López, M., & García-Castro, R. (2022). LOT : An industrial oriented ontology engineering framework. Engineering Applications Of Artificial Intelligence, 111, 104755. https://doi.org/10.1016/j.engappai.2022.104755
- Visalli, M., Schlich, P., Mahieu, B., Thomas, A., Weber, M., & Guichard, E. (2022). First steps towards FAIRization of product-focused sensory data. Food Quality And Preference, 104, 104765. https://doi.org/10.1016/j.foodqual.2022.104765
- Weber, M., Buche, P., Ibanescu, L., Dervaux, S., Guillemin, H., Cufi, J., Visalli, M., Guichard, E., & Pénicaud, C. (2023). PO2/TransformON, an ontology for data integration on food, feed, bioproducts and biowaste engineering. Npj Science Of Food, 7(1), 47. https://doi.org/10.1038/s41538-023-00221-2
- Weber, M., Duclos, F., Guillemin, H., Dervaux, S., Cufi, J., & Visalli, M. (2025). ConsomON, an ontology for structuring knowledge and modeling consumer food behavior. Joint Ontology Workshops (JOWO) - Episode XI: The Sicilian Summer under the Etna, co-located with the 15th International Conference on Formal Ontology in Information Systems (FOIS 2025), IFOW- Integrated Food Ontology Workshop, Sep 2025, Catania, Italy. https://hal.inrae.fr/hal-05146634v2
- Wilkinson, M. D., Dumontier, M., Aalbersberg, I. J., Appleton, G., Axton, M., Baak, A., Blomberg, N., Boiten, J.-W., Da Silva Santos, L. B., Bourne, P. E., Bouwman, J., Brookes, A. J., Clark, T., Crosas, M., Dillo, I., Dumon, O., Edmunds, S., Evelo, C. T., Finkers, R., … Mons, B. (2016). The FAIR Guiding Principles for scientific data management and stewardship. Scientific Data, 3(1), 160018. https://doi.org/10.1038/sdata.2016.18
Pièces jointes
Pas de document complémentaire pour cet articleStatistiques de l'article
 Vues: 550
Vues: 550
Téléchargements
 XML: 22
XML: 22
 PDF: 103
PDF: 103

