Conception d’un support de prises de vue et d’une chaine d’analyse d’images pour l’estimation rapide de la surface foliaire sur feuilles détachées
Résumé
La surface foliaire est une variable centrale pour de nombreuses recherches en sciences du végétal. Son estimation peut être réalisée in situ de manière non destructive via une proxy ou une télédétection, ou de manière destructive avec l'utilisation d'un planimètre où chaque feuille est scannée. Les méthodes non destructives sont généralement plus rapides mais présentent des biais et incertitudes plus importantes. Au contraire, l'approche par planimètre est plus précise mais très chronophage et demande un matériel spécifique coûteux et difficilement transportable. Dans le cadre du suivi de la croissance foliaire de la pomme de terre, nous avons effectué l'estimation rapide de la surface foliaire de feuilles détachées par imagerie quantitative. Nous avons conçu un support de prise de vue avec une chaîne d’analyse d’images codée sous ImageJ et Python qui permet de mesurer les surfaces foliaires et d’être utilisée en conditions extérieures (champ) ou semi-contrôlées (hangar, tunnel, serre …). Notre méthode peut être facilement adaptée à différentes espèces végétales, au niveau d’études concernant le couvert végétal, la plante ou l’organe, et nécessitant la mesure rapide et peu onéreuse de surfaces foliaires. Nous présentons ici les résultats issus de notre dispositif avec un échantillon de 683 plantes de pomme de terre, dont deux variétés, prélevées et mesurées au cours de trois saisons de culture. La validation de ce dispositif s’est effectuée avec des données acquises simultanément au planimètre et montre une très bonne corrélation des surfaces mesurées par les deux méthodes. Notre dispositif présente de plus, un gain de temps très intéressant et un faible coût (quelques dizaines d’euros), même s’il sous-estime un peu la surface foliaire (17 %).
Introduction
La mesure de la variable « surface foliaire » représente un intérêt dans de nombreuses expérimentations végétales. En effet, elle permet de quantifier la croissance et le développement des plantes et du couvert végétal et également d’établir des indices tel que le LAI (Leaf Area Index) pour quantifier la densité du couvert végétal (Daughtry et al.,1990 ; Jonckheere et al., 2003).
Actuellement plusieurs approches sont possibles pour la mesure de la surface foliaire. Il y a d’abord les méthodes dites « directes » parce qu’elles concernent réellement une mesure de surface foliaire (destructive ou non), telles que la planimétrie des feuilles avec l’utilisation d’un planimètre (passage de la feuille sur une source lumineuse et mesure de la surface à l’aide d’une caméra), le scanner à plat (mesure à l’aide d’un logiciel d’analyse d’images du scan) ou la mesure manuelle des feuilles (mesure de la longueur, largeur des feuilles à l’aide d’une règle ou d’un pied à coulisse). Il y a ensuite les méthodes dites « indirectes » et non destructives à l’aide de données de lumière réfléchie (réflectance) pour différentes longueurs d’ondes du spectre lumineux, généralement rouge et infra-rouge. Ces données peuvent être corrélées à la biomasse végétale et donc à la l’estimation de la surface foliaire. Ces méthodes non destructives permettent notamment de calculer des indices de végétation tels que le NDVI « Normalized Difference Vegetation Index » ou l’EVI2 « Enhanced Vegetation Index 2 » (Jiang et al., 2008). L’ensemble de ces techniques est très chronophage, demande du matériel coûteux et peut nécessiter des connaissances, au niveau de l’utilisation des capteurs, pas toujours disponibles dans certaines équipes d’expérimentation.
Dans le cadre du suivi de la croissance foliaire de la pomme de terre, nous avons développé une approche qui comprend le développement d’un support ad hoc et la mise au point d’une chaîne de traitement d’images pour extraire les surfaces foliaires. Cette méthode a été éprouvée et validée lors de quatre années d’expérimentation au champ sur des couverts de pomme de terre. Elle permet de réaliser des mesures de surface foliaire plus rapidement qu’avec un planimètre et avec un matériel peu coûteux, facilement déployable sur plusieurs sites et utilisable pour de grandes séries de mesures.
Cet article détaille la conception et la validation expérimentale de la méthode proposée, ainsi qu’une analyse comparative de ses performances par rapport aux méthodes standards de mesure de surface foliaire, notamment l’utilisation d’un planimètre.
Objectifs du dispositif
Afin de pallier les limites des méthodes traditionnelles, souvent coûteuses, chronophages et peu adaptées au terrain, nous avons défini des objectifs spécifiques pour le développement d’un dispositif de mesure foliaire, en insistant sur la rapidité, la portabilité, la simplicité d’utilisation et l’automatisation complète de la chaîne de traitement des images.
Les surfaces obtenues ont été comparées avec celle d’un planimètre nous servant de mesures de références avec pour objectif d’avoir une bonne corrélation (coefficient de détermination > à 90) et une différence de surface inférieure à 20 % entre les 2 méthodes sur l’ensemble de la gamme des surfaces mesurées.
Le support de prise de vue devait être :
- Léger et facilement transportable.
- Comporter l’ensemble des repères permettant la segmentation et le calibrage de l’image.
- Être suffisamment grand pour pouvoir mettre un grand nombre de feuilles sans chevauchement de celles-ci.
- Facilement transférable à d’autres utilisateurs et à d’autres cultures.
- Utilisable avec tous types d’appareils numériques de prise de vue (compacts, reflex, smartphone…).
- La chaîne de traitement des images devait être automatisée via le développement de scripts et comporter plusieurs étapes :
- Le calibrage de l’image pour la mesure des surfaces.
- La sélection de la région d’intérêt (ROI).
- La segmentation des feuilles.
- La mesure et l’enregistrement des résultats dans un fichier de sortie.
- La création d’une image de contrôle des zones sélectionnées.
Principe de la méthode et matériels utilisés
Description du support
Après avoir testé plusieurs fonds, nous avons retenu un support en PVC expansé de 10 mm d’épaisseur. Cette matière est rigide, résistante, facile à nettoyer et légère pour le transport (Figure 1). Nous l’avons choisie dans la gamme de blanc plutôt mât pour éviter le phénomène de brillance et ainsi faciliter l’extraction des feuilles.

Figure 1. Support pour la prise de vue standardisée
Le support proposé se compose (Figure 1) :
- D’une zone d’identification (1), permettant d’inscrire les informations de l’échantillon pris en photo et/ou de positionner un code barre ou un QR code utilisable pour un renommage automatique de la photo et facilitant le suivi de l’échantillon.
- D’une zone de calibration d’échelle (2), comportant une référence pour calculer la surface réelle des objets mesurés.
- D’une charte de balance des blancs (3), pour pouvoir, si besoin, uniformiser les couleurs entre toutes les séries d’images.
- D’une zone d’intérêt (ROI) (4), délimitée par des repères à l’intérieur de laquelle on positionne les feuilles à mesurer.
- De marqueurs visuels :
- (5) ronds bleus de 15 mm de diamètre pour le découpage automatique des images avec les scripts Fiji (Figure 2a),
- (6) marqueur de type « ArUco » (Garrido-Jurado et al., 2014) qui sont des marqueurs de références carrés synthétiques, composés d'une large bordure noire et d'une matrice binaire interne qui détermine son identifiant (ID). Ils sont utilisés ici pour le découpage de la zone d’intérêt et le redressement automatique des images dans les scripts Python (Figure 2b).
a) Ronds bleus de 15 mm de diamètre ; b) marqueurs « AruCo » avec 4 identifiants utilisés (ID-0, ID-1, ID-2, ID-3 du dictionnaire ArUco 4X4_50, dans la librairie OpenCv) 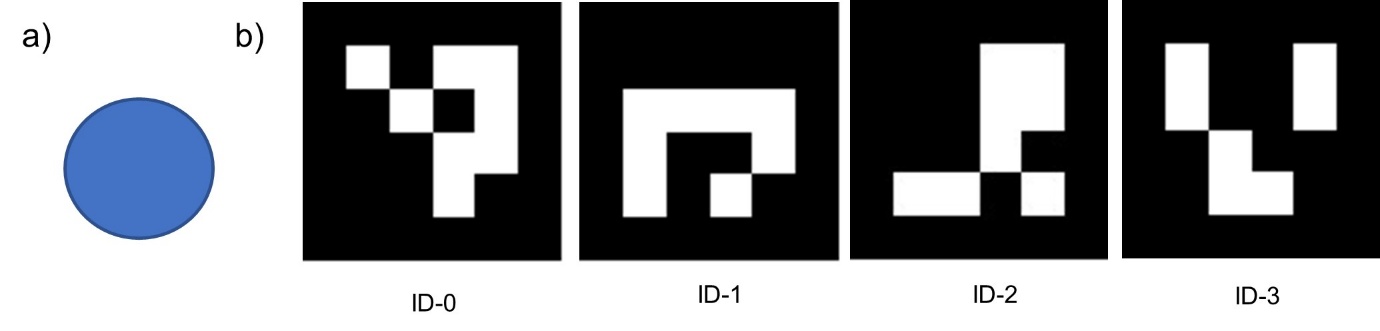
Figure 2. Marqueurs visuels
Acquisition des images
L’acquisition des images peut se faire à partir d’un appareil photo numérique reflex, compact ou smartphone avec une définition suffisante, si possible supérieure à 2000 x 1000 pixels.
La prise de vue s’effectue par le dessus et le plus près possible du nadir (Figure 3). En effet, une inclinaison de l’appareil photo peut provoquer une déformation de perspective et donc des feuilles. Cette prise de vue est effectuée soit directement à « main levée » en tenant l’appareil photo au-dessus du support (Figure 3a, 3b) soit à l’aide d’une perche de prise de vue avec un système de déclenchement à distance (Figure 3c) et un appareil maintenu à la verticale par un système de stabilisation automatique (Figure 3d).
a) et b) prise de vue à main levée du support ; c) perche de prise de vue ; d) stabilisateur automatique pour une prise de vue par-dessus (vers le nadir)
Figure 3. Acquisition des images
Pour les conditions de prise de vue, celles sous abri sont privilégiées (hangars, ailes techniques). Au champ, il faudra privilégier une zone protégée des intempéries, pluie et vent, et des changements trop violents de conditions lumineuses, notamment pour éviter les zones d’ombre trop importantes ou les zones de surbrillances. Même si le traitement d’image est robuste, les variations trop importantes de lumière peuvent modifier le traitement, en particulier pour la partie de segmentation des feuilles par seuillage. Ici, le seuil permettant de séparer les pixels correspondant à une feuille du fond est choisi manuellement après un changement d’espace de couleurs de l’image.
Implémentation d’une chaîne automatisée de traitement des images
Choix des logiciels et du langage de programmation
L’objectif principal du dispositif présenté ici était d’associer ce support à une série d’algorithmes implémentés sous Fiji (Schneider et al., 2012 ; Schindelin et al., 2012 ; Rueden et al., 2017) et Python (Python Software Foundation, 2023), deux solutions logicielles libres, pour la mesure rapide de la surface foliaire sur un grand nombre d’images.
Le choix de l’utilisation Fiji ou Python dépend de l’utilisateur. En effet, Fiji va nous permettre une automatisation des tâches dans le traitement des images avec une interface utilisateur conviviale pour l'écriture et l'exécution de macros, même par des utilisateurs qui ne sont pas des programmeurs. En parallèle, nous avons souhaité développer également l’ensemble du traitement en langage Python pour au moins trois raisons :
- Son large écosystème de bibliothèques. En effet, Python dispose de bibliothèques dédiées au traitement d'images, dont les plus populaires sont OpenCV, scikit-image et Pillow (PIL). Ces bibliothèques offrent une gamme étendue de fonctionnalités pour manipuler, analyser et traiter des images. C’est un langage qui est reconnu pour sa syntaxe claire et sa facilité d'apprentissage avec une communauté très active. À titre d’exemple, la bibliothèque OpenCv inclut des fonctionnalités pour détecter et reconnaître des marqueurs comme les ArUco (Figure 2) que nous avons rajoutés sur notre support pour découper et redresser l’image ; ces fonctions ne sont pas disponibles sous Fiji.
- Le traitement des images sous Python sera moins sensible aux variations importantes de luminosité (pas de marqueurs visuels colorés) et aux variations d’angles de prises de vue (présence de la fonction de redressement de l’image).
- De plus, le traitement sera beaucoup plus rapide avec Python (6 000 images traitées à l’heure contre 500 avec Fiji avec un ordinateur possédant un processeur Intel(R) Core(TM) i7-12700K 3.60 GHz et 64 Go de RAM).
Les deux scripts, Fiji (Leaf_Area_Support_2023.ijm) et Python (Code_Python_Canopy_2024.py) sont disponibles sur la forge MIA-INRAE à l’adresse suivante : https://forge.inrae.fr/stephane.jumel/support-and-associated-algorithms-for-measuring-leaf-area
Les différentes étapes de traitement pour le calcul de la surface foliaire
Une image visible classique est codée en trois canaux de couleur Red, Green, Blue (RGB). Une même image peut être représentée dans un autre espace de couleur tridimensionnel comme l’espace CIELab (ou Lab) qui représente les couleurs selon la luminance (clair/foncé, axe L), et deux axes a et b représentant la couleur sur des axes vert – rouge et bleu – jaune respectivement (Gonzalez & Woods, 2017). L’utilisation de l’espace Lab permet notamment de mieux séparer la couleur de la luminosité, ce qui est utile lorsque l’éclairage varie dans l’image. Les pixels de l’image correspondant aux feuilles peuvent être obtenus en appliquant des seuils sur les histogrammes de chaque canal de l’espace de couleur. Ici, on considérera des couleurs dans l’espace Lab et des seuils par défaut que nous avons définis comme optimaux dans nos conditions, qui pourront être modifiés par l’utilisateur, suivant ses besoins, dans la boîte de dialogue au lancement du script.
Les différentes étapes du traitement sont (Figure 4) :
a) L’extraction de la zone d’intérêt
- Avec Fiji :
- Ouverture de l’image originale et changement d’espace de couleur de RGB à Lab Seuillage sur le canal « b » pour segmenter uniquement les ronds bleus des 4 angles de la ROI (valeur du seuillage à 105, Tableau 1).
- Conversion en masque et utilisation de la fonction « Analyse particle » de Fiji pour sélectionner uniquement les masques des 4 ronds bleus.
- Extraction des coordonnées du centroïde de chaque rond et traçage d’un rectangle à partir de celles-ci.
- La fonction « Crop » de Fiji nous permettra de découper l’image de la ROI.
- Avec Python :
- Détection des 4 marqueurs ArUco présents dans l’image à l’aide de la fonction ArucoDetector d’OpenCV.
- Extraction des coins et des identifiants des marqueurs afin de découper l’image à chaque angle de la région d’intérêt.
- Redressement de l’image avec la fonction « cv.getPerspectiveTransform » pour compenser les distorsions de perspectives éventuelles dues à l’inclinaison de l’appareil lors de la prise de vue.
b) Le calibrage des mesures
- Avec Fiji :
- Ouverture de l’image originale.
- Changement d’espace de couleur (RGB à Lab) puis réalisation d’un seuillage sur le canal « a » pour segmenter uniquement la mire rouge de l’image (valeur du seuillage à 153, Tableau 1).
- Conversion en masque et utilisation de la fonction « Analyse particle » pour sélectionner uniquement le masque de la mire.
- Extraction des caractéristiques (longueur et largeur) pour paramétrer l’échelle de mesure.
- Avec Python :
- Utilisation des valeurs connues en cm (et renseignées dans la boite de dialogue au démarrage du script) entre les différents marqueurs de référence ArUco, présents à chaque angle de la région d’intérêt.
c) La segmentation des feuilles
- Avec Fiji :
- Ouverture de l’image de la ROI et changement d’espace de couleur (RGB à Lab) puis réalisation d’un seuillage sur le canal « a » pour segmenter uniquement les feuilles présentes de la ROI (valeur du seuillage à 123, Tableau 1).
- Conversion en masque et utilisation de la fonction « Analyse Particles » pour sélectionner uniquement le masque des feuilles.
- Calcul de la surface du masque (feuilles) à partir de la fonction « Measure » de Fiji et enregistrement automatique des résultats sous un fichier Excel que l’on génère dans le dossier de sortie.
- Avec Python :
- Utilisation des fonctions d’OpenCV « cv.cvtColor » pour le changement d’espace de couleur et « cv.threshold » pour le seuillage et le masque des feuilles (valeur 123 par défaut, Tableau 1)
d) La création d’images de contrôle
- Avec Fiji :
- Création de l’image de contrôle à partir de la fonction « imageCalculator ». Cette fonction effectue la superposition de l’image du contour du masque des feuilles avec l’image originale de la ROI nous montrant ainsi les zones sélectionnées lors du traitement.
- Avec Python :
- Enregistrement du masque des feuilles à l’aide de la fonction « cv.imwrite » pour le contrôle des zones sélectionnées lors du seuillage.
e) L’enregistrement des résultats dans un fichier Excel
- Avec Fiji :
- Enregistrement des résultats de surface foliaire en cm² dans un fichier Excel, à partir d’un plug-in Fiji nommé « Read and Write Excel » que l’on exécute à la fin de la macro.
- Avec Python :
- Utilisation de la bibliothèque « pandas » afin de générer le tableau Excel (.xlsx) des résultats de surfaces foliaires.
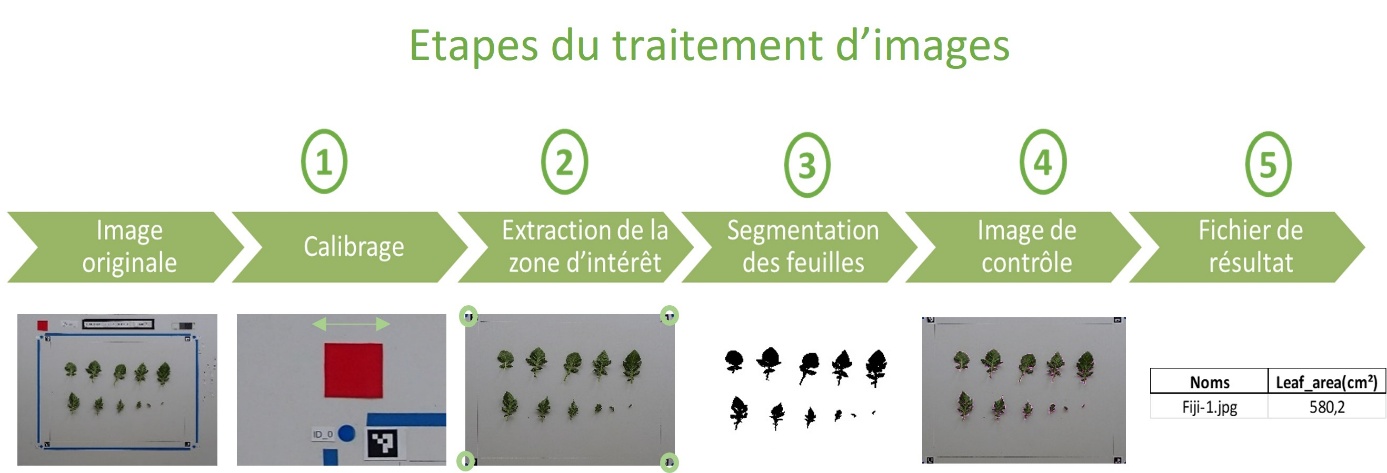
Figure 4. Étapes de la chaîne de traitement d’images
Utilisation des scripts
Après l’ensemble des acquisitions, les images sont stockées à l’emplacement de votre choix et sont ensuite analysées directement depuis ces dossiers par un script d’analyse d’images (format .ijm) utilisant le logiciel ImageJ/FIJI ou un script utilisant Python (format .py). Ces scripts permettent, après leur exécution, d’analyser sans aucune intervention de l’utilisateur l’ensemble des images et de générer automatiquement un tableau Excel des données de surfaces.
- Avec Fiji, lancez le script après l’ouverture du logiciel. Au lancement du script, une boîte de dialogue s’ouvre pour demander le chemin du dossier où sont stockées les images. Quelques paramètres sont à renseigner comme le format des fichiers images, la largeur de la zone d’intérêt et la valeur de seuillage (Figure 5).
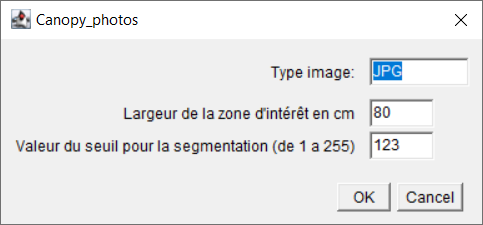
Figure 5. Boite de dialogue au lancement de la macro Fiji
- Avec Python, on indique la largeur et la hauteur de la zone d’intérêt correspondant à la distance entre les coins de chaque marqueur ArUco, puis le chemin du dossier contenant les images à traiter (Figure 6). Les autres paramètres sont à changer, si besoin, directement dans le script (Tableau 1).
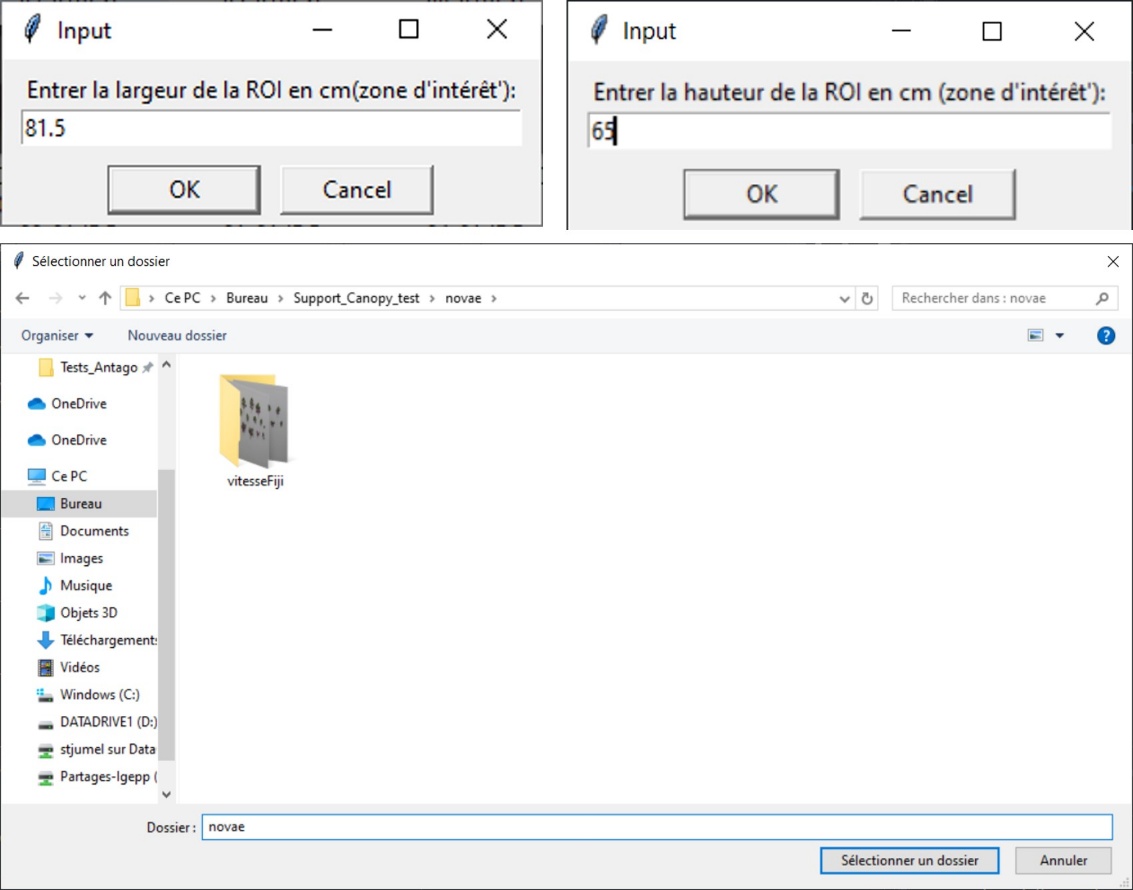
Figure 6. Boite de dialogue au lancement du script en Python
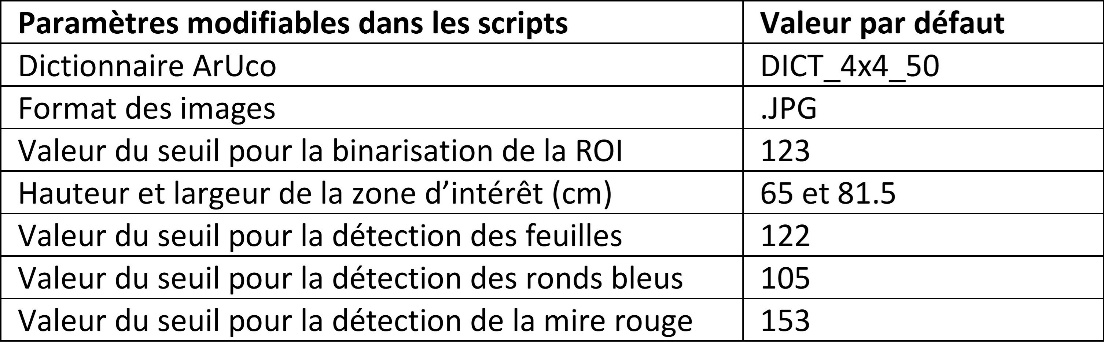
Tableau 1. Paramètres modifiables
Validation expérimentale par comparaison des surfaces obtenues entre notre approche et des mesures au planimètre
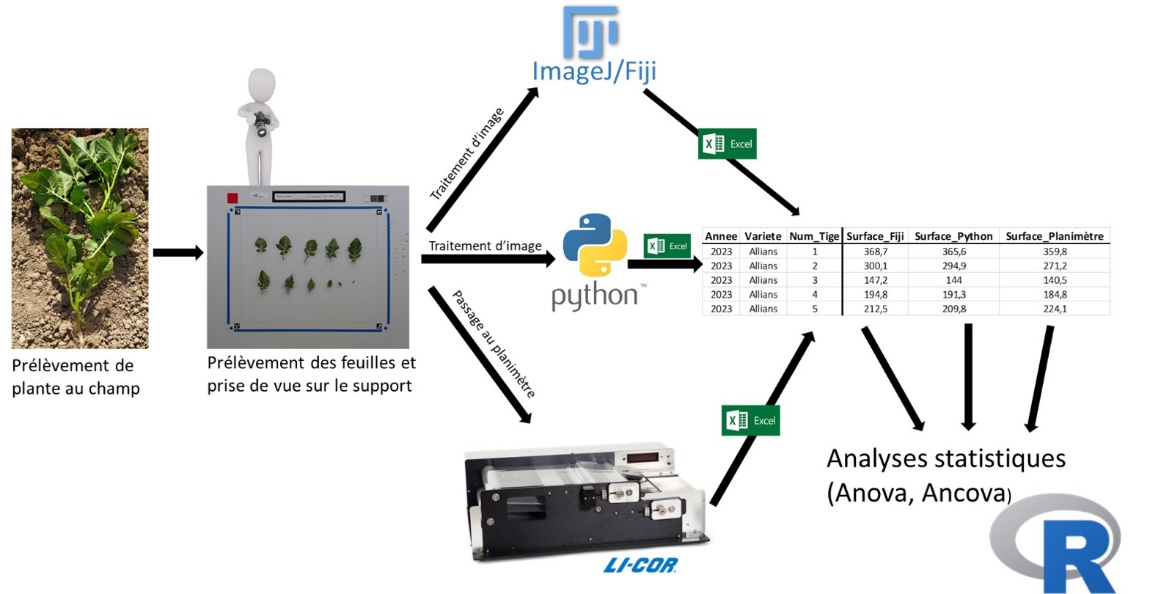
Figure 7. Schéma de la validation expérimentale de la méthode
Mesure de la surface foliaire par notre approche
Notre méthode de mesure de la surface foliaire a été validée en comparant les mesures qu’elle fournit à celles obtenues par un planimètre (Figure 7). Cette validation a été réalisée sur des feuilles de pomme de terre provenant de deux variétés, « Passion » et « Allians », respectivement cultivées pendant trois et une saison de culture sur le site INRAE de Le Rheu (48°06′10″N 1°47′39″W). Dans chaque cas, trois plantes ont été prélevées deux fois par semaine de la levée jusqu’à la maturité (floraison et stabilisation de la fermeture du couvert). Au total 683 plantes comportant entre 2 et 12 tiges ont été phénotypées. Après le prélèvement des plantes, pour chaque tige de chaque plante les feuilles ont été séparées (et non les folioles) et déposées, le plus à plat possible, dans la zone d’intérêt sur le support de prise de vue (Figure 8). Lorsque les feuilles d’une tige étaient trop importantes et ne pouvaient pas toutes être installées sur le support sans chevauchement, on réalisait deux prises de vue. Pour chaque tige, une image dans le visible a été acquise avec un appareil photo compact (Olympus TG4), un appareil photo reflex (Nikon D5300) ou plusieurs modèles de smartphone suivant l’opérateur. Pour chaque image acquise, la surface foliaire de chaque tige a été estimée en utilisant la méthode implémentée en ImageJ Macro Language (sur 3 années d’expérimentation) et celle en Python (sur 1 année d’expérimentation).

Figure 8. Disposition des feuilles sur le support
Mesure de la surface foliaire par planimètre
Après l’acquisition des images sur le support de prise de vue, la surface de chaque feuille a été mesurée avec un planimètre. Le planimètre est un appareil utilisé pour des mesures précises de surface foliaire. Pour cette étude, nous avons travaillé avec le modèle LI-3100C de chez LI-COR (Figure 9) ayant les caractéristiques suivantes :
- Résolution réglable de 0,1 mm2 ou 1 mm2.
- Mesure toutes les feuilles avec des largeurs de 1 cm à 25 cm.
- Système de courroie avec rouleaux presseurs réglables pour aplatir les feuilles courbées ou mesurer les feuilles épaisses jusqu'à 2,5 mm.
Le planimètre mesure la surface de chaque échantillon déposé entre des bandes plastiques transparentes via la surface projetée de l’objet. Les feuilles passent au-dessus d’une source lumineuse et sont détectées par la caméra localisée sur le boîtier arrière de l’appareil (Figure 9). Tant que l’utilisateur ne réinitialise pas la mesure, l’appareil cumule la surface de tous les objets passés, ce qui permet d’obtenir facilement la surface foliaire de chaque tige après passage de chaque feuille. Une fois la mesure réalisée, le notateur note la surface mesurée et affichée par l’appareil dans un tableau de données.

Figure 9. Passage des feuilles au planimètre (Li-Cor LI-3100C)
Comparaison des différentes méthodes de mesure de la surface foliaire
Validation de la corrélation entre les mesures du script Fiji et le planimètre sur 3 années d’expérimentations
La comparaison visuelle des distributions des surfaces foliaires obtenues par notre méthode et le planimètre suggère une bonne proximité que ce soit au cours de la saison de culture ou entre les années (Figures 10a, 10b, 10c).
Afin d’évaluer le biais entre notre méthode de mesure de la surface foliaire et l’utilisation d’un planimètre, nous avons réalisé une régression linéaire en considérant les trois années d’expérimentation (2021-22-23) (Figure 10d). Cette analyse statistique confirme une très bonne corrélation avec un coefficient de détermination (R²) de 0.98 et une pente de 0.83 (Figure10d, droite rouge par rapport à la 1re bissectrice si x=y en bleu) montrant une légère sous-estimation de 17 % des mesures de surfaces issues de notre dispositif par rapport au planimètre. Cette sous-estimation peut s’expliquer en partie par l’aplatissement des feuilles lors du passage au planimètre, contrairement aux images prises sur le plateau de prise de vue.
a), b), c) Box plots comparatifs des surfaces foliaires mesurées à plusieurs dates pour les 3 années (entre 8 et 12 dates de mesures par année) ; d) Régression linéaire entre les données issues du planimètre et celles de la chaîne de traitement avec notre support pour l’ensemble des données sur 3 années d’expérimentations.Figure 10. Comparaison des mesures obtenues par notre méthode et par un planimètre
Comparaison des mesures obtenues par les implémentations des scripts Fiji, Python et le planimètre
Afin de comparer les deux implémentations Fiji et Python entre elles (Figure 11) nous avons considéré l’ensemble des données de l’année 2023 (101 plantes, 498 tiges sur 2 variétés). Nous avons comparé les mesures obtenues par les implémentations Fiji versus Python en réalisant une régression linéaire pour comparer les données obtenues avec l’utilisation des deux scripts sur les mêmes images. Cette analyse montre une parfaite adéquation des surfaces mesurées et ne permet pas de mettre en évidence de différence significatives entre les deux méthodes dans nos conditions expérimentales. (R²~1, pente =1, Figure 11).
De plus, nous avons testé d'éventuels effets de l'implémentation utilisée (Fiji/Python), de la variété de pomme de terre utilisée (Allians/Passion) et de l'interaction entre ces deux facteurs sur le biais entre les mesures obtenues par notre méthode et celles issues du planimètre en réalisant une ANCOVA. Cette analyse n'a pas permis de mettre en évidence un quelconque effet significatif de la variété ou de l'algorithme utilisé sur la pente ni sur l'ordonnée à l'origine du biais.
Toutes les analyses statistiques ont été réalisée avec le logiciel R (R Core Team, 2021).
Régression entre les données de surfaces issues du script Fiji et celles du script Python pour la saison 2023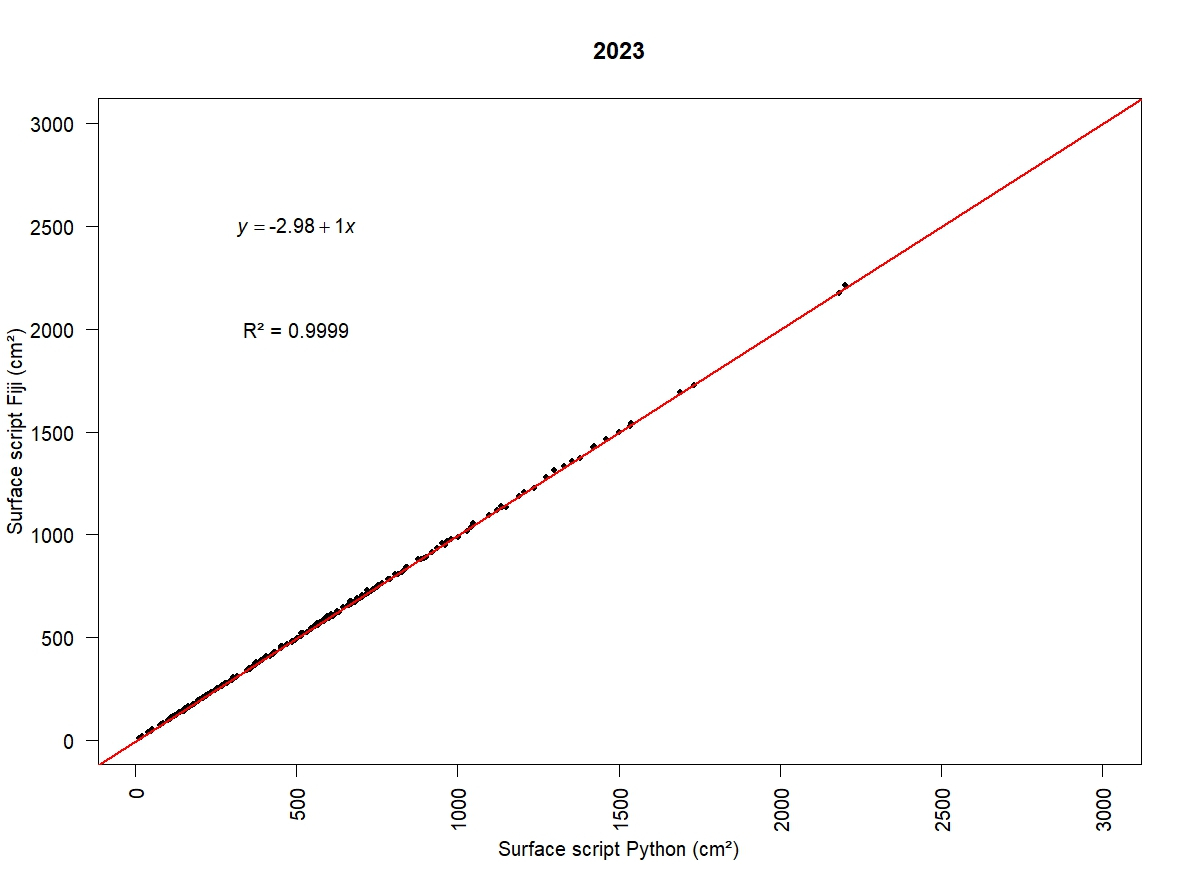
Figure 11. Comparaison entre les données issues du script Fiji et Python
Conclusion
La méthode d'estimation de la surface foliaire couplant acquisition et analyse d’images, que nous avons développée, se distingue par sa rapidité et son faible coût. Contrairement à un planimètre dont le prix avoisine 14 000 euros HT, notre dispositif repose sur un simple support à quelques dizaines d’euros, un appareil photo ou un smartphone couramment disponible dans les laboratoires, ainsi que des scripts basés sur des logiciels libres.
Notre solution permet de traiter un volume d’échantillons bien plus important qu’un planimètre, connu pour être chronophage. L’acquisition d’images atteint une cadence de 25 à 30 prises de vue par heure, tandis que notre chaîne d’analyse sous Python permet de traiter environ 100 images par minute. Bien que Fiji soit une alternative plus accessible pour les utilisateurs non familiers avec Python, il reste nettement plus lent pour le traitement de grands jeux de données.
Ce dispositif a été utilisé durant quatre années d’expérimentation dans le cadre du projet Casdar RT Canopy, permettant le suivi de la croissance foliaire de la pomme de terre sur quatre sites expérimentaux et la mesure de 1 000 plantes dans des sites expérimentaux dépourvus de planimètre. Malgré une sous-estimation de la surface foliaire de 10 à 17 % par rapport à un planimètre (Figures 10a, 10b, 10c), notre méthode reste précise et fiable. Le biais étant stable, il peut être corrigé a posteriori pour permettre une comparaison directe avec les mesures du planimètre. Il serait intéressant d’évaluer ce biais sur d’autres variétés ou espèces et de le confronter à d’autres méthodes, notamment non destructives.
L’un des principaux atouts de notre dispositif réside dans sa flexibilité. Il peut être adapté à d'autres espèces et organes (tubercules, racines, tiges) en ajustant quelques paramètres du traitement d’images. Il peut également être modifié pour mesurer d’autres traits phénotypiques, tels que la longueur, la hauteur des plantes ou des caractéristiques topologiques (angles, niveaux d’insertion des feuilles…).
Perspectives d’amélioration
Le traitement d’images représente un axe majeur d’amélioration. Sous Fiji, l’intégration de marqueurs visuels, tels que les marqueurs ArUco, permettrait de corriger automatiquement les distorsions liées à la prise de vue et offrirait une alternative plus fiable que les marqueurs utilisés avec Fiji (ici des ronds bleus). De plus, l’utilisation de méthodes de segmentation plus avancées, notamment des algorithmes de Deep Learning, pourrait améliorer la précision des mesures par rapport au seuillage simple utilisé actuellement.
À court terme, nous envisageons également de concevoir un support pliable et modulable, adaptable à différentes configurations expérimentales. La possibilité de stocker les images sur le long terme garantit une traçabilité optimale et permet d’effectuer de nouvelles analyses a posteriori, un avantage que n’offre pas le planimètre.
Enfin, nous prévoyons d’étendre et de valider notre méthode sur d’autres espèces végétales dans le cadre de futurs projets, afin d’optimiser encore davantage la caractérisation des couverts végétaux et des plantes étudiées.
Remerciements
Nous tenons à remercier l’ensemble du personnel des sites d’expérimentation de la FN3PT ayant participé au test du dispositif, Loïc Daniel pour la mise en place de l’essai sur le site de Le Rheu, Gwenaelle Deniot pour avoir accepté d’être sur une photo d’illustration et Houda Braham de Sciences Impact pour son aide à la rédaction. Ce travail a été financé par le Ministère de l’Agriculture via le projet Casdar Recherche Technologique «Canopy» (20ART1568974).
Références
- Daughtry, C., Kustas, W., Moran, Pinter, P., Jackson, R., Brown, P., Nichols, W., & Gay, L. (1990). Spectral estimates of net radiation and soil heat flux. Remote Sensing Of Environ-ment, 32(2 3), 111 124. https://doi.org/10.1016/0034-4257(90)90012-b
- Garrido-Jurado, S., Muñoz-Salinas, R., Madrid-Cuevas, F., & Marín-Jiménez, M. (2014). Automatic generation and detection of highly reliable fiducial markers under occlusion. Pat-tern Recognition, 47(6), 2280 2292. https://doi.org/10.1016/j.patcog.2014.01.005
- Gonzalez, R., & Woods, R. (2017). Digital Image Processing. Pearson eLibrary. https://elibrary.pearson.de/book/99.150005/9781292223070
- Jiang, Z., Huete, A., Didan, K., & Miura, T. (2008). Development of a two-band enhanced vegetation index without a blue band. Remote Sensing Of Environment, 112(10), 3833 3845. https://doi.org/10.1016/j.rse.2008.06.006
- Jonckheere, I., Fleck, S., Nackaerts, K., Muys, B., Coppin, P., Weiss, M., & Baret, F. (2003). Review of methods for in situ leaf area index determination. Agricultural And Forest Meteoro-logy, 121(1 2), 19 35. https://doi.org/10.1016/j.agrformet.2003.08.027
- Python Software Foundation (2023). Python Language Reference (version 3.10). https://www.python.org
- R Core Team (2021). R : A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. https://www.R-project.org/
- Rueden, C. T., Schindelin, J., Hiner, M. C., DeZonia, B. E., Walter, A. E., Arena, E. T., & Eli-ceiri, K. W. (2017). ImageJ2 : ImageJ for the next generation of scientific image data. BMC Bioinformatics, 18(1). https://doi.org/10.1186/s12859-017-1934-z
- Schindelin, J., Arganda-Carreras, I., Frise, E., Kaynig, V., Longair, M., Pietzsch, T., Preibisch, S., Rueden, C., Saalfeld, S., Schmid, B., Tinevez, J., White, D. J., Hartenstein, V., Eliceiri, K., Tomancak, P., & Cardona, A. (2012). Fiji : an open-source platform for biological-image ana-lysis. Nature Methods, 9(7), 676 682. https://doi.org/10.1038/nmeth.2019
- Schneider, C. A., Rasband, W. S., & Eliceiri, K. W. (2012). NIH Image to ImageJ : 25 years of image analysis. Nature Methods, 9(7), 671 675. https://doi.org/10.1038/nmeth.2089
Pièces jointes
Pas de document complémentaire pour cet articleStatistiques de l'article
 Vues: 493
Vues: 493
Téléchargements
 PDF: 91
PDF: 91
 XML: 24
XML: 24

